María Ripoli, Guillermo Giovambattista, Julio De Lúea, Fernando
Dulout,
Centro de Investigaciones en Genética Básica y Aplicada (CIGEBA),
60 y 118, CC 296, 1900 La Plata, Argentina, ggiovam@fcv.medvet.unlp.edu.ar.
Fernando Labarta, José Echenique, Marcelo Sánchez, Edgardo Carrizo,
Facultad de Ciencias Agrarias. Universidad Nacional de Jujuy.
Bolivia 1239. 4600 San Salvador de Jujuy
Recepcion originales 17 de Mayo de 1999
Formation of a foundation herd of Argentine Creole Cattle for milk production.
Effect on six loci gene frequencies.
Key words: Creole cattle, Selection, Genetics Markers, Milk production.
With the purpose to establish a foundation group to carry out a dairy improvement
plan for Argentine Creole cattle, thirty-eight dams and one sire belonging to
this breed were selected. In order to evaluate such selection effect on six
loci, polymorphisms of b-lactoglobulin, growth hormone, F13A, BoLA-DRB3,
BoLA-DYA, and the microsatellite D23S5 were characterized in the
selected population (38 animals), as well as in unselected population (78 animals).
The characterization of b-lactoglobulin, growth hormone, F13A, BoLA-DRB3,
BoLA-DYA alleles was carried out through the techniques of PCR-RFLP,
while the D23S5 variants were characterized by PCR-sequencing gel methodology.
These results revealed a significant increase of b-lactoglobulin B variant and
non significant increase in D23S5 B variant. Whereas no significant difterences
between the selected and unselected population were observed for gene frequencies
of growth hormone F13A, BoLA-DRB3, BoLA-DYA.
The observed differences could be consequence of selection eftect. Family studies
would be nessesary to confirm that the observed differencies were consequence
of association between these molecular markers with quantitative trait loci
(QTLs) for milk production.
Palabras Claves: Ganado criollo, selcción marcadores genéticos,producción
de leche.
Con el objetivo de establecer un plantel base de ganado Bovino Criollo Argentino
para producción lechera, se seleccionaron treinta y ocho vientres y un
toro pertenecientes a esta raza. Para poder evaluar el efecto de la selección
sobre las frecuencias génicas, se caracterizaron los polimorfismos presentes
en seis loci: b-lactoglobulina, hormona de crecimiento, F13A, BoLA-DRB3,
BoLA-DYA, y en el microsatélite D23S5, tanto en la población
seleccionada (38 animales), como en el rebaño no seleccionado (78 animales).
La caracterización de b-lactoglobulina, hormona de crecimiento, F13A,
BoLA-DRB3, BoLA-DYA se llevó a cabo a través de la técnica
de PCR-RFLP, mientras que las variantes del microsatélite D23S5
se caracterizaron mediante PCR-electroforesis en geles de secuenciación.
Los resultados evidenciaron un aumento significativo de la variante B de b-lactoglobulina,
y un incremento no significativo de la variante B del microsatélite D23S5.
Por otra parte las frecuencias génicas de los loci hormona de crecimiento,
F13A, BoLA-DRB3, BoLA-DYA, no mostraron diferencias significativas entre
la población seleccionada y la no seleccionada. Las diferencias observadas
podrían ser consecuencia del efecto de la selección. Sin embargo,
sería necesario realizar estudios familiares para confirmar que las diferencias
observadas serían consecuencia de la asociación entre los marcadores
moleculares analizados con loci de caracteres cuantitativos (QTLs) para producción
lechera.
Numerosos estudios realizados durante las últimas cuatro décadas,
han puesto de manifiesto correlaciones estadísticamente significativas
entre marcadores genéticos y caracteres de producción lechera.
Dichos estudios han sido enfocados hacia el análisis de loci candidatos
y/o hacia el rastreo del genoma mediante el uso simultáneo de un gran
número de marcadores moleculares altamente polimórficos, tales
como los microsatélites (Zwiauer, 1980; Beattie,
1994; Haley, 1995; Georges & Andersson,
1996; Giovambattista et al., 1998). Se ha definido
a los loci candidatos como aquellos genes que por su función biológica
podrían participar en la expresión de un carácter cuantitativo.
Pueden mencionarse a modo de ejemplo las proteínas de la leche, la prolactina
(PRL), la hormona de crecimiento (GH), etc.
Existen evidencias bien documentadas que permiten correlacionar algunas de las
variantes de las proteínas de la leche (k-caseína, b-caseína,
aS1-caseína, aS2-caseína,
a-lactoglobulina y b-lactoglubulina) con el rendimiento y la composición
del producto lácteo obtenido (e.g. McLean et
al., 1984; Boland et al., 1992; Lin
et al, 1986; McLean y Schaar, 1989; Ng-Kwai-Hang
et al, 1984, 1990; Cowan et al,
1990). También se han asociado las variantes alélicas de diversas
hormonas (PRL y GH) con caracteres de produción lechera
(Cowan et al., 1990 ; Hoj et al.,
1993; Yao et al., 1996a).
Además, cabe mencionar que varios genes del Complejo Principal de Histocompatibilidad
pueden tener influencia, en forma directa o indirecta, sobre los niveles de
producción (Batra et al., 1989; Beever
et al, 1990; Stear et al, 1988; Weigel
et al,, 1990 ; Ostergärd et al, 1989;
Mejdell et al, 1993, 1994a, 1994b).
Por otra parte, en los últimos años, se han encontrado posibles
asociaciones entre distintos microsatélites y caracteres de producción
lechera (Georges et al, 1993; Ron et al, 1994;
Georges et al, 1995; Spelman et al
1996 ; Ashwell et al, 1996; Vilkki
et al 1997). Por lo tanto, el análisis del polimorfismo de
los loci candidatos y de los microsatélites podría constituir
una información útil para los programas de selección de
ganado lechero.
La mayoría de los estudios basados en el análisis de marcadores
genéticos se han llevado a cabo principalmente en razas seleccionadas
para distintos fines productivos, donde la variabilidad se ve reducida en gran
medida. Es decir, se están analizando las consecuencias de un largo proceso
selectivo y no el proceso en sí mismo.
El ganado bovino Criollo Argentino (ACc), definido como el descendiente puro
y directo de los vacunos introducidos por los conquistadores españoles
en el territorio argentino (Primo, 1992), presenta un alto
grado de variabilidad genética por no haber soportado una alta presión
de selección artificial. Además, como consecuencia de muchos años
de selección natural, el ganado ACc se ha adaptado a una amplia variedad
de condiciones ambientales, desarrollando una gran variabilidad fenotípica,
altos niveles de longevidad y fertilidad, además de una alta resistencia
a enfermedades subtropicales (Sal Paz, 1986; Hansen,
1994).
El objetivo del presente trabajo consistió en evaluar el efecto de la
primera etapa de selección de bovinos Criollos Argentinos Lecheros sobre
las frecuencias génicas de seis marcadores genéticos. Dos de ellos,
GH y b-lactoglubulina (b-LG), se han asociado con producción
lechera, mientras que el BoLA-DRB3 ha sido correlacionado con la respuesta
inmune y por lo tanto podría afectar, en forma indirecta, los niveles
de producción. Los loci restantes, BoLA-DYA, F13A y D23S5
(MGTG7) no han sido asociados hasta el momento con cantidad y calidad
composicional de la leche. De esta manera se amplió el estudio realizado
en los mismos rebaños por Ripoli et al (1999),
en el que se estimó el efecto de la selección sobre las frecuencias
génicas de los loci k-caseína, aS1-caseína
y prolactina
Poblaciones Estudiadas
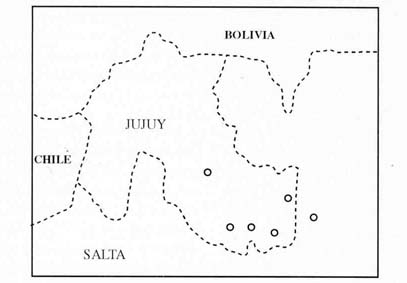
Rebaño General de animales no seleccionados. El rebaño
general analizado correspondió a la población de la estación
Zootécnica Subtropical ARROYO DEL MEDIO (EEAM), ubicada en el monte chaqueño
de transición, en el sudeste de la provincia de Jujuy, Argentina (Sánchez
Mera, 1989). Dicha estación recibió animales puros de la raza
ACc provenientes de las localidades de El Pongo, Maimará, Lavayén,
San Juan de Dios, Palma Sola en la provincia de Jujuy, y Anta en la provincia
de Salta (Figura 1). Actualmente, la población de la EEAM cuenta con
un plantel de aproximadamente 150 animales.
 |
| Figura 1: Ubicación geográfica de las localidades de Las Animas (1), Maimará (2), San Antonio (3), Estación Experimental El Remate (4), El Pongo (5), Lavallén (6), San Juan de Dios (7), Estación Zootécnica Subtropical Arroyo del Medio (8), Anta (Salta) (9), y Palma Sola (10). |
Selección y adquisición de animales para el proyecto lechero.Con
el objetivo de establecer un plantel base para la selección de bovinos
Criollos lecheros se seleccionaron 32 vientres y un toro de la EEAM. Por otra
parte, fueron adquiridos 6
animales a productores locales de la provincia de Jujuy (4 de Las Animas, Dpto.
de Tilcara y 2 de La Cabana, Dpto. de San Antonio). Actualmente, este plantel
se encuentra localizado en la Estación Experimental El Remate (EEER).
Las hembras provenientes de las Estaciones Experimentales fueron seleccionadas
según los siguientes criterios: pesos de los terneros al destete logrados
por los vientres durante su vida productiva, biotipo, antecedentes genealógicos
y otros antecedentes lecheros obtenidos en las Estaciones Experimentales.
El sistema empleado para la compra de las vacas a productores consistió
en la búsqueda de datos en distintos puestos de hacienda de los departamentos
de San Antonio y Tilcara, con la finalidad de individualizar animales sujetos
a ordeña y seleccionar algunas vacas y vaquillas para incorporarlas al
plantel lechero. En este caso la selección se basó en los antecedentes
suministrados por sus criadores, en el fenotipo, conformación y suspensión
de las ubres; tamaño, orientación y pigmentación de los
pezones; ausencia de pezones supernumerarios y temperamento de los animales
(Carrizo, 1995).
Metodología Experimental
Se obtuvieron 116 muestras de sangre periférica de bovinos de raza ACc
pertenecientes a las poblaciones de EEAM (78 animales) y de EEER (38 animales).
El aislamiento del ADN se llevó a cabo mediante la técnica de
extracción con DNA ZOL, (Gibco BRL - life Technologies, Grand Island,N.Y,USA).
Técnicas de tipificación de los marcadores genéticos
analizados: Hormona de crecimiento (GH): Para amplificar un fragmento
de 404 pb correspondiente al quinto exón del gen GH se utilizó
la técnica descrita por Yao et al. (1996a).
Dicho fragmento posee el sitio de restricción polimórfico para
la enzima Alu I que define los alelos A y B.
Alícuotas de los productos de amplificación (12 ml) fueron digeridos
con la enzima de restricción Alu 1 (2,5 U) (Promega, Madison,
WI). Los fragmentos de restricción fueron resueltos en geles de poliacrilamida
6% (19:1) - TBE 1x, teñidos con bromuro de etidio y visualizados con
luz UV.
b-lactoglobulina (b-LG): La caracterización
del locus b-LG se realizó mediante la técnica de PCR-RFLP descrita
por Medrano y Aguilar-Córdoba (1990). Esta metodología
permitió amplificar un fragmento de 247 pb correspondiente al cuarto
exón, en donde se encuentra el sitio de restricción polimórfico
para la enzima Hae III que permite definir los alelos A y B.
Alícuotas de los productos de amplificación (12 ml) fueron digeridos
con la enzima de restricción Hae III, (2,5 U) (Promega, Madison,
WI). Los fragmentos de restricción fueron resueltos en geles de poliacrilamida
6% (19:1) - TBE Ix, teñidos con bromuro de etidio y visualizados con
luz UV.
Subunidad A del factor de coagulación sanguínea (F13A):
La caracterización del tercer exón del locus F13A se realizó
mediante el método heminested-PCR-ACRS descrito por Park
et al. (1995). Esta técnica permite identificar las variantes
alélicas de este locus mediante la creación de un sitio de restricción
artificial, utilizando un cebador que posee en el extremo 3' una base
no complementaria al ADN molde y que se ubica junto a la base polimórfica.
Alícuotas de los productos de amplificación (12 ml) fueron digeridos
con la enzima de restricción Taq I (2,5 U) (Promega, Madison,
WI). Los productos de amplificación fueron resueltos en geles de poliacrilamida
6 % (19:1) - TBE 1x, teñidos con bromuro de etidio y observados con luz
UV.
BoLA-DYA: La caracterización del segundo exón del
gen BoLA-DYA se realizó mediante el método PCR-ACRS, el
cual crea un sitio de restricción artificial para la enzima Hind
III (Van der Poel et al 1990).
Alícuotas de los productos de amplificación (12 ml) fueron digeridos
en forma simultánea con las enzima de restricción Hind
III (2,5 U) y Hha I (2,5 U) (Promega, Madison, WI), para lo cual se utilizó
el buffer universal MULTI-CORE (Promega, Madison, WI).
Los productos de amplificación fueron resueltos en geles de poliacrilamida
6 % (19:1) - 1x TBE, teñidos con bromuro de etidio y visualizados con
luz UV.
BoLA-DRB3: La caracterización de los alelos del locus BoLA-DRB3
se realizó mediante la técnica heminested-PCR-RFLP descrita por
Lewin et al (1992), la que permite amplificar un fragmento
de 284 pb correspondiente al segundo exón (sitio de reconocimiento del
antígeno ARS) del gen.
Alícuotas de los productos de amplificación fueron digeridas,
por separado, con las enzimas de restricción Rsa I, Bsty
I y Hae III (2,5 U) (Promega, Madison, WI).
Los fragmentos de restricción fueron resueltos en geles de poliacrilamida
al 6 u 8 % (19:1) - TBE 1x, coloreados con bromuro de etidio y visualizados
con luz UV.
Los alelos correspondientes a este locus fueron definidos mediante la combinación
de los patrones obtenidos para las tres enzimas de restricción, según
la nomenclatura propuesta por van Eijk et al.,( 1992).
MGTG7 (D23S5): Para amplificar el fragmento correspondiente al
microsatélite MGTG7 (288 - 310 pb) se utilizaron los cebadores
descritos por Bates et al., (1996).
Los alelos fueron resueltos en geles desnaturalizantes de poliacrilamida al
6% (19:1) - TBE Ix, corridos a 50 watts durante 4 horas y revelados mediante
la técnica de tinción con plata.
Metodología Estadística
Las frecuencias génicas y genotípicas se calcularon para cada
uno de los loci analizados en el rebaño seleccionado para producción
lechera (EEER) y en el rebaño general (EEAM).
El equilibrio de Hardy Weinberg se calculó a través del índice
F1S. El valor de significancia del índice
F1S se estimó mediante el Test de Probabilidad
o el Test Exacto de Fisher descrito por Raymond y Rousset (1995).
La comparación de las frecuencias genotípicas observadas en los
dos rebaños se llevó a cabo a través de tablas de contingencia,
cuando las limitaciones del método lo permitieron.
La comparación entre las frecuencias génicas observadas en los
dos rebaños fue realizada a través del análisis de Chi-Cuadrado,
del método binomial de comparación de dos proporciones y del índice
FST modificado para comparar pares de poblaciones
(Zar, 1984; Nei, 1987; Raymond
y Rousset, 1995). Estos análisis se llevaron a cabo mediante la utilización
de los programas Statgraphics, Plus, versión 3 (Manugistic, Inc., Rockville,
MA, USA). Quatro (Borland International, USA) y Genepop (Raymond y Rousset,
1995) respectivamente. El valor significancia del índice FST
se estimó mediante el Test de Probabilidad o el Test Exacto de Fisher
descrito por Raymond y Rousset, (1995).
Los niveles de diversidad genética se estimaron a través del número
de alelos detectados (na) y del índice
de heterocigosidad esperada (he). Además se calculó
la heterocigosidad promedio (He) para los dos
grupos, considerando tanto a los seis loci analizados en el presente trabajo,
asi como a los genes aS1-caseína, k-caseína
y PRL (Ripoli et al., 1999).
La comparación entre las He estimadas
para cada plantel se realizó mediante el método "t-Student"
(Nei, 1987). Las varianzas intra e interlocus de las He
se calcularon según las fórmulas propuestas por Nei
(1987).
En el cuadro 1 se detallan los valores de las frecuencias génicas y
genotípicas estimadas para los loci de b-LG, GH y F13A.
Dichos valores fueron calculados para el rebaño seleccionado de la EEER
y el rebaño general de la EEAM. Las frecuencias génicas calculadas
para el locus BoLA-DYA y para el microsatelite D23S5, se detallan
en el cuadro II; mientras que las frecuencias génicas estimadas para
el locus BoLA-DRB3 se describen en el cuadro III.
La estimación del índice FIS evidenció
que todos los loci analizados en ambas poblaciones se hallaban en equilibrio
de Hardy-Weinberg, a excepción del microsatelite D23S5 que se encontró
en desequilibrio en el rodeo EEAM (Cuadros 1, 2, 3). Cuando se calculó
el índice FIS en foma conjunta para todos los loci,
las dos poblaciones se encontraron en equilibrio (c2EEER
= 17,800; p = 0,599; c2EEEM=
17,0; p = 0,651).
La comparación de las frecuencias génicas y genotípicas
correspondientes a los 2 grupos analizados puso de manifiesto que:
b-LG: En el rebaño general las frecuencias génicas para
las variantes A y B fueron similares, mientras que en el grupo seleccionado
se observó un aumento del alelo B con respecto a la población
original. Estas diferencias se encontraron en el límite de significación
(c2 = 3,950; p = 0,0469; Z = 1,945; 0,05 <
p < 0,10) (Cuadro 1), lo que fue confirmado mediante el índice FST
(Cuadro 4). La comparación de las frecuencias genotípicas correspondientes
a ambos rodeos no evidenció diferencias significativas (c2
= 4,830; p = 0,089).
| Cuadro 1: Frecuencias génicas y genotípicas observadas
para los loci de b-lactoglobulina, hormona de crecimiento y subunidad A
del factor de coagulación sanguínea XIII en el rebaño
general y el grupo seleccionado. Table 1: Geneic and genotype frecuencies observed for loci of B-lactoglobuline groeth hormon and sun-unit a of blood clotting in the herd as well as the selected group. |
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
GH: En el rebaño general y en el seleccionado las frecuencias
génicas para las variantes A y B fueron similares. Las diferencias entre
los dos rodeos no fueron significativas (c2
- 0,02; p = 0,878; Z = 0,131; p > 0,50) (Cuadro 1), lo que fue confirmado
mediante el índice FST (Cuadro 4). La
comparación de las frecuencias genotípicas correspondientes a
ambos rebaños no evidenció diferencias significativas (c2
= 1,10; p = 0,576).
F13A: Las frecuencias génicas para las variantes B y b
fueron similares en los dos rebaños, no observándose diferencias
significativas entre las frecuencias génicas ( c2-
0,81; p = 0,368; Z = 0,679; p = 0,50) (Cuadro 1), 1o que fue confirmado mediante
el índice FST (Cuadro 4). La comparación
de las frecuencias genotípicas correspondientes a ambos rebaños
tampoco evidenció diferencias significativas (c2
= 1,01; p =0,603).
BoLA-DYA: Las cuatro variantes alélicas reportadas en bovinos
se detectaron en ambos rebaños. El alelo G1G2 fue el más abundante
en las dos poblaciones, con una frecuencia superior al 60%, la variante G1A2
se detectó en ambos grupos con frecuencia del orden del 25%, mientras
que los alelos A1A2 y A1G2 fueron detectadas en las dos poblaciones con una
frecuencia inferior al 5 % (Cuadro 2). Las diferencias entre las frecuencias
génicas estimadas para ambos rodeos resultaron ser no significativas
(c2 = 1,45; 0,99 > p > 0,975), lo que
fue confirmado mediante el índice FST
(Cuadro 4). La comparación de las frecuencias genotípicas correspondientes
a ambas poblaciones tampoco evidenció diferencias significativas (c2
= 1,370; p = 0,712).
| Cuadro 2: Frecuencias génicas observadas para el locus BoLA-DYA
y para el microsatélite D23S5 en el rebaño general y el grup
seleccionado. Table 2: Gentic frequencis observed for locus Bola-Dya and for the microsatellite D2355 in the herdand selected group. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Cuadro 3: Frecuencias génicas observadas para el locus de BoLA-DRB3
en el rebaño general y el grupo seleccionado. Table 3: Gentic frequencis observed for locus Bola-Dya and for the microsatellite D2355 in the herdand selected group. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
D23S5: Las mismas vanantes alélicas se detectaron en ambos
rebaños. Las frecuencias génicas estimadas para dichos alelos
fueron distintas en las dos poblaciones, sin embargo las diferencias no fueron
significativas (c2 = 5,23; p = 0,073) (Cuadro
2), lo que fue confirmado mediante el índice FST
(Cuadro 4). La comparación de las frecuencias genotípicas correspondientes
a ambas poblaciones tampoco evidenció diferencias significativas (c2
= 7,50; p = 0,186).
BoLA-DRB3: Se detectaron un total de quince variantes alélicas
en la EEAM y doce en la EEER. Los alelos DRB3.2*1, DRB3.2*7 y
DRB3.2*23 sólo se encontraron, con frecuencias muy bajas, en la
población de la EEAM. El alelo DRB3.2*15 fue mayoritario en ambos
rebaños con una frecuencia del orden del 20%. Los alelos DRB3.2*10,
DRB3.2*18, DRB3.2*20 y DRB3.2*22 presentaron frecuencias considerables
(entre 7% y el 15%) en las dos poblaciones. El resto de las variantes alélicas
también fueron semejantes en ambos rebaños, con frecuencias menores
al 10%, a excepción del alelo DRB3.2*5 cuya frecuencia fue del
orden del 10% en la EEAM y del 2% en la EEER. Las diferencias entre las frecuencias
génicas estimadas para ambos rebaños a través del índice
FST resultaron ser no significativas (Cuadro 4).
| Cuadro 4: Valores estimados del índice FST, con
su respectiva probabilidad y error estándar, para los loci analizados. Table 4: Index timate values with its probality and standard error, for analiged loci. |
||||||||||||||||||||||||||||||||
|
||||||||||||||||||||||||||||||||
| Cuadro 5: Estimación del na, he y ES por
loci en cada una de las poblaciones. Table 5: na, he y ES estimation per loci on lach sampling. |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Baker y Manwell (1980), realizaron la primera clasificación
de las razas bovinas basada en el análisis de marcadores genéticos.
A este primer trabajo le sucedieron estudios realizados por otros autores, como
por ejemplo, Medjugorac et al., (1994); MacHugh
et al, (1994, 1997). El análisis de las relaciones entre las
razas es posible si se asume que los marcadores genéticos utilizados
son selectivamente neutros, y que las poblaciones están en equilibrio
bajo deriva y migración, por lo que la similitud entre las razas refleja
el ancestro común. Por lo tanto los procesos de selección no habrían
modificado las frecuencias génicas originales, las que serían
consecuencia de la historia particular de cada raza, de los movimientos migratorios
y de la distribución geográfica actual (Baker y
Manwell, 1980; Medjugorac et al., 1994). Es por
esta razón que las distintas razas han podido ser agrupadas en base a
sus frecuencias génicas, coincidiendo dichas agrupaciones con las realizadas
mediante datos históricos y morfológicos.
Por otra parte, estudios cronológicos han evaluado el efecto de los procesos
de selección sobre las frecuencias génicas. Para cumplir con dicho
objetivo, se compararon las frecuencias alélicas de toros utilizados
en inseminación artificial durante las décadas del 50 y del 60,
con toros de la misma población utilizados durante la década del
80. Estos trabajos evidenciaron diferencias significativas entre los dos grupos
analizados, a modo de ejemplo se pueden mencionar los genes de k-caseína
y de ornitina descarboxilasa (Velmala et al, 1993;
Yao et al., 1996b).
En 1999, Ripoli et al estudiaron las consecuencias
de la formación de un plantel base de criollos lecheros sobre las frecuencias
génicas de los loci k-caseína, as1-caseína
y prolactina. Dicho estudio evidenció diferencias significativas en las
frecuencias génicas del locus de as1-caseína.
En el presente trabajo, se extendió el análisis a los loci GH,
b-LG, F13A, BoLA-DRB3, BoLA-DYA y al microsatélite
D23S5.
La comparación entre las frecuencias génicas del rodeo seleccionado
de la EEER y del rodeo general de la EEAM, mostró diferencias en el límite
de significación en el locus b-LG. Mientras que en el resto de
los genes las diferencias observadas no fueron significativas. Este hecho indicaría
que la formación del plantel base resultó en una modificación
de las frecuencias génicas en los loci b-LG y D23S5. Sin
embargo estas diferencias no fueron tan marcadas como las reportadas para el
locus de as1-caseína (Ripoli
et al, 1999).
Las frecuencias génicas de una población pueden verse modificadas
por factores tales como endogamia, deriva génica, selección, etc.
Las diferencias observadas en el presente estudio, no serían consecuencia
de la acción del primer factor mencionado. Esto puede justificarse mediante
el análisis de los pedigrí, ya que ambos rebaños presentaron
niveles equivalentes de endogamia.
Por otra parte, la selección sólo puede actuar si la sustitución
de un alelo por otro implica una variación en la adaptación del
animal (Bovenhuis y Weller, 1994). Un gran número de
trabajos han estudiado el efecto del gen b-LG sobre los caracteres de
producción lechera en razas europeas altamente seleccionadas (Giovambattista
et al, 1998). A nivel poblacional, estudios del comportamiento productivo,
han puesto de manifiesto que la variante A está asociada con un mayor
nivel de b-lactoglobulina y proteínas totales del suero de la leche,
y con un menor contenido de caseínas y grasa (Boland et
al, 1992). Por otra parte, los animales con genotipo b-LG BB
tienen un menor contenido de b-lactoglobulina y un mayor contenido de caseínas,
proteínas totales y grasa en la leche, lo que resulta en mejores propiedades
para la producción de queso (Tong et al, 1993).
Además este genotipo ha sido correlacionado con un mayor rendimiento
en litros de leche con respecto al genotipo AA, habiéndose observado
para el genotipo AB un rendimiento intermedio (Jadot et al,
1992). En coincidencia con lo observado en las razas europeas, la selección
de animales para producción lechera (EEER) produjo un aumento de la variante
B que se encontró en el límite de significación.
La expresión diferencial de los alelos A y B de b-LG ha sido
observada en diferentes poblaciones de ganado bovino lechero (Aschaffenburg
y Drewry, 1955; Cerbulis y Farrel, 1975; McLean
et al., 1984; Kroeker et al, 1985; Ng-Kwai-Hang
et al, 1987; Hill, 1993; Lum et
al, 1997). Esto es debido a la presencia de sustituciones nucleotídicas,
entre ambas variantes alélicas, en la región promotora de b-LG
que modifican la afinidad de los factores de transcripción (por ejemplo:
AP-2) y por lo tanto los niveles de expresión. En los animales heterocigotos
aproximadamente el 60% de la b-lactoglobulina corresponde a la variante A, mientras
que el 40% restante está contituída por la variante B (Graml
et al., 1989; Wilkins et al, 1995;Lum
et al., 1997).
En cuanto al gen GH, Lee et al (1996), estudiaron
el sitio de restricción polimorfía) Alu I (exón
5) en un rebaño seleccionado y en otro control de la raza Holstein. A
pesar que en el primer grupo, el genotipo Alu -/- estaba asociado con
una disminución en el valor de cría para la producción
lechera, no observaron diferencias entre las frecuencias génicas de ambos
grupos. Por otra parte, Hoj et al. (1993), agruparon
bovinos pertenecientes a las razas Danesa y Noruega Rojas en dos grupos, según
fuera alta o baja la producción de grasa en leche. Los animales fueron
tipificados para el polimorfismo I/D (región 3') y para el sitio de restricción
Msp I (intrón 3). Los resultados obtenidos indicarían que
la selección sobre el carácter grasa en leche habría incrementado
las frecuencias de los alelos D y Msp I (-). Finalmente, Yao
et al. (1996a) analizaron el polimorfismo del gen GH en toros
Holstein, a través de la técnica de SSCP (Single Strand Conformation
Polymorphism). Dos de los fragmentos variables evidenciaron asociaciones
significativas entre las variantes de GH con mayor producción
de leche, grasa y proteínas.
Sin embargo, en el presente trabajo las frecuencias génicas correspondientes
al locus GH (Alu I +/-) no evidenciaron diferencias significativas
entre el grupo seleccionado y el rebaño general. La ausencia de diferencias
podría ser consecuencia de distintos factores. Si tenemos en cuenta que
Yao et al.(1996a) reportaron la presencia de dos variantes
de SSCP por cada uno de los alelos Msp I (+) y Alu I (+), este
hecho podría ser una de las causas de los resultados obtenidos.
Por otra parte, dado que el ligamiento entre el QTL (Quantitative
Trait Loci) y el marcador genético es incompleto, podrían
existir en la población diferentes haplotipos marcador-QTL. Así
por ejemplo, el alelo GH A podría estar ligado al alelo de alta
producción del QTL en algunos animales, mientras que en otros
estaría ligado a la variante de baja productividad del QTL. Por
lo tanto, los efectos estimados del QTL sobre el rasgo cuantitativo se
verán sesgados por efecto de la recombinación. Por último,
cabe mencionar que no necesariamente en todas las razas, se tienen que presentar
las mismas asociaciones entre un marcador y un QTL.
Así por ejemplo, en la raza Danesa Roja la variante Msp I (-)
fue asociada con mayor producción lechera, mientras que en la raza Holstein
este alelo se asoció con baja producción de leche (Hoj
et al., 1993; Yao et al., 1996a).
El Complejo Principal de Histo-compatibilidad (denominado Bovine Lymphocyte
Antigen, BoLA), ocupa un rol central en la respuesta inmune. Este complejo es
de gran importancia para la adaptación del individuo al medio, por lo
que influye directa o indirectamente sobre los caracteres de producción.
Numerosos estudios han demostrado la asociación entre el BOLA
y caracteres de importancia fisiológica como fertilidad (Ostergärd
et al., 1989; Mejdell et al., 1993, 1994
a,b), caracteres de producción, parámetros de crecimiento
(Batra et al., 1989; Beever et al.,
1990; Stear et al., 1988; Weigel
et al., 1990), susceptibilidad o resistencia a enfermedades, como
por ejemplo leucosis bovina, mastitis y parásitos intestinales (Lewin
y Bernoco, 1986; Bernoco y Lewin, 1989; Lewin,
1989; Lundén et al., 1990; Schook
et al., 1991; Zanotti et al., 1995; 1996).
Estos estudios revelaron la importancia del análisis de estos marcadores,
tanto para el estudio de la resistencia a enfermedades, como para los caracteres
de producción (Lewin et al., 1991), ya que numerosas
evidencias han demostrado que los animales portadores de las enfermedades antes
mencionadas presentan una importante disminución en la producción
(Yang et al., 1993). Entre los genes más estudiados
se pueden mencionar el locus de Clase II BoLA-DRB3 y entre los
loci de Clase I, el BoLA-A.
En el presente trabajo se analizaron tres loci pertenecientes al Complejo
Mayor de Histocompatibilidad: BoLA-DRB3, BoLA-DYA y D23S5,
También fue incluido en el estudio el gen F13A el que se encuentra,
al igual que dicho complejo, en el BTA23. Los resultados obtenidos no evidenciaron
diferencias significativas. Sin embargo, el valor estimado para el microsatelite
D23S5 se encontró cercano al límite de significación.
La ausencia de diferencias para los loci BoLA-DYA y F13A entre
ambos rebaños, podría ser consecuencia de su ubicación
en el cromosoma 23. Como se mencionó anteriormente los loci del BoLA
(Clase I y II) que han sido asociados con caracteres de importancia económica
se encuentran en la región 2 del BTA23, mientras que el BoLA-DYA
se encuentra cercano al centrómero, y el F13A próximo al
telómero. Por lo tanto la ausencia de diferencias podría deberse
a que hasta el momento, en ninguna de estas dos regiones han sido reportados
QTLs, y por otra parte a la gran distancia (cM) existente entre el BoLA-DYA
y el F13A con respecto a los loci de Clase I y II.
Con respecto al locus BoLA-DRB3 y al microsatelite D23S5, la formación
del plantel base de criollos lecheros sólo modificó las frecuencias
génicas de éste último, a pesar de encontrarse ambos estrechamente
ligados. Este hecho podría deberse al gran número de alelos detectados
para el BoLA-DRB3 con respecto al D23S5.
A su vez, el número reducido de animales que conforman el plantel seleccionado
hace que existan muchas clases representadas por pocos individuos, lo que dificulta
la comparación de los dos grupos analizados.
Con respecto a la heterocigocidad, existen evidencias que en razas europeas
altamente seleccionadas hay pérdida de variabilidad (e.g. MacHugh
et al., 1997). En la población de la EEER no se produjo disminución
de la heterocigocidad con respecto al rebaño general, al menos en esta
primera etapa de selección. Sería de interés analizar las
siguientes generaciones seleccionadas para producción lechera, para evaluar
si los niveles de diversidad genética se mantienen a lo largo del proceso
de selección.
Finalmente, la existencia de correlación entre las variantes alélicas
del locus b-LG, así como para el locus aS1-caseína
reportado anteriormente (Ripoli et al., 1999), con
una mayor producción lechera en la raza ACc, debería confirmarse
aumentando el número de animales caracterizados y a través de
estudios familiares, en los que se relacionen dichos marcadores genéticos
con los caracteres cuantitativos. De esta manera, podría determinarse
la presencia de haplotipos característicos para dicha raza y confirmar
que las diferencias observadas en el presente trabajo se deberían al
proceso de selección y no a un error de muestreo.
ASCHAFFENBURG R. y J. DREWRY. 1955. Ocurrence of different
betalactoglobulins in cow's milk. Nature (Lond.) 176 : 218.
ASHWELL, M.S., C.E. REXROAD JR, R.H.MILLER y P.M. VANRADEN.
1996. Mapping economic trait loci for somatic cell score in Holtein cattle using
micosatellites markers and selective genotyping. Animal Genetics 27 :235-242.
BAKER, A.C.M. and MANWELL, C. 1980. Chemical classification
of cattle. I. Breed groups. Anim. Blood Grps Biochem. Genet. 11:127-150.
BATES, S., C. PETERSON-KNABE, T. HOLM, H. VAN HAERINGEN, K.
LANCE, J. ZIEGLE, D. HEYEN, Y. DA Y H. LEWIN. 1996. Exclusion probabilities
of 22 bovine microsatellites markers in fluorescent multiplexes for automated
parentage verification. Proceeding of the XXV International Society for Animal
Genetics 28.
BATRA, T. R., A. J. LEE, R. S. GAVORA y M. J. STEAR. 1989.
Class I alleles of the bovine major histocompatibility system and their association
with economic traits. J. Dairy Sci. 72: 2115-2124.
BEATTIE, C. 1994. Livestoek genome maps. TIG 10 : 334 - 338.
BEEVER, J.E., P.D. GEORGE, R.L. FERNANDO, C.T. STORMONT y H.A.
LEWIN. 1990. Association between genetic markers and growth and carcass traits
in a paternal half-sib family of Angus cattle. J. Anim. Sci. 68 : 337 - 344.
BERNOCO, D. y H.A. LEWIN. 1989. The bovine lymphocyte antigen
(BoLA) system: Importance andrelatioship to disease in cattle. Rev. Bras. Genet.
12(3) : 107 - 122.
BOLAND, M.L, J.P. HILL y L.K. CREAMER. 1992. Genetic manipulation
of milk proteins & its consequences for the dairy industry. Australian Biotechnology
2(6) : 355 - 360.
BOVENHUIS, H. and WELLER, J.I. 1994. Mapping and analysis of
dairy cattle quantitative trait loci by maximum likelihood methodology using
milk protein genes as genetic markers. Genetics 137: 267-280.
CARRIZO, E.N. 1995. Informe de avance del proyecto SeCTER/UNju
A-49.1. Mejoramiento del Bovino Criollo como Productor de Leche en la Región
Subtropical del NOA.
CERBULIS, J. y H. M. FARREL, JR. 1975. Compositions of milks
of dairy cattle. I. Protein, lactose, and fat contents and distribution of protein
fraction. J. Dairy Sci. 58 : 817.
COWAN, C. M., M. R. DENTINE, R. L. AX y L. A. SCCHULER. 1990.
Structural variation around prolactin gene linked to quantitative traits in
an elite Holstein sire family. Theor. Appl. Genet. 79: 577 - 582.
GEORGES M., D. NIELSEN, M. MACKINNON, A. MISHRA, R. OKINOTO,
A.T. PASQUINO, L.S. SARGEANT, A. SORENSEN, M.R. STEELE y X. ZHAO. 1995. Mapping
quantitative trait loci controlling milk production in dairy cattle by exploting
progeny testing. Genetics 139 : 907-920.
GEORGES M., M. LARTHROP, A. B. DIETZ, A. LEFORT, F. LIBERT,
A. MISHRA, D. NIELSEN, L. S. SARGEANT, M. R. STEELE, X. ZHAO, H. LEIPOLD y J.
E: WOMACK. 1993. Microsatellite mapping of the gene causing weaver disease in
cattle will allow the study of an associated QTL. Proc. Nati. Acad. Sci. 90:
1058-1062.
GEORGES, M.y L. ANDERSSON (1996) Livestock genomics comes of
age. Genome Research 907-921.
GIOVAMBATTISTA G., M.V. RIPOLI, P. PERAL GARCÍA, J.C.
DE LUGA, EN. DULOUT. 1998. Estado actual del mapeo de QTLS para producción
lechera. Agrosur. 26: 78-96.
GRAML, R., G. WEISS, J. BUCHBERGER y F. PIRCHNER. 1989.
Different rates of synthesis of whey protein and casein by alleles of the beta-lactoglobulin
and alpha-s1-casein locus in
cattle. Genet. Sel. Evol. 21 : 547.
HALE Y, C. S. 1995. Livestoek QTLs-bringing home the bacon?.
TIG 11(12) : 488 - 492.
HANSEN,E.W. 1994. Ganadería bovina de raza criolla en
el noroeste argentino. Universidad Nacional de Jujuy, San Salvador de Jujuy,
Argentina. 150 pp.
HILL, J.P. 1993. The relationship between (-lactoglobulin phenotypes
and milk composition in New Zealand dairy cattle. J. Dairy Sel. 76 : 281.
HOJ, S., M. FREDHOLM, N. LARSEN y V.H. NIELSEN. 1993. Growth
hormone gene polymorphism associated with selection for milk fat production
in lines of cattle. Animal Genetics 24 : 91-96.
JADOT, M., J. LALOUX, A. BURNY y R. KETTMANN. 1992. Detection
of bovine -lactoglobulin genomic variants by the polymerase chain reaction method
and molecular hybridization. Animal Genetics 23(1) : 77 - 80.
KROEKER, E. M., K. F. NG-KWAI-HANG, J. F. HAYES y J. E. MOXLEY.
1985. Effect of (-lactoglobulin variant and enviromental factors on variation
in the detailed composition of bovine milk serum proteins. J. Dairy Sci. 68
: 1637.
LEE, B.K., G.F. LIN, B.A. CROOKER, M.P. MURTAUGH, L.B. HANSEN
y H. CHESTER-JONES. 1996. Association of somatotropin (BST) gene polymorphism
at the 5th exon with selection for milk yield in Holstein cows. Domestic Animal
Endocrinology 13(4) : 373-381.
LEWIN, H. A. y D. BERNOCO. 1986. Evidence for BoLA linked resistance
and susceptibility to subclinical progression of bovine leukaemia virus infection.
Animal Genetics 17 : 197 - 207.
LEWIN, H.A. 1989. Disease resintance and immune response genes
in cattle: strategies for their detection and evidence of their existence. J.
Dairy Sci. 72: 1334 - 1348.
LEWIN H. A., P. A. CLAMP, J. E. BEEVER y L. B. SCHOOK.1991.
Mapping genes for resistance to infectious diseases. In "Gene mapping :
Techniques and applications" (L. B. Schook, H. A. Lewin, and D. G. McLaren,
Eds.), pp. 283-303, Dekker, New York.
LEWIN, H.A., K. SCHMITT, R. HUBERT, M.J.T. VANELTKy N.ARNHEIM.
1992. Close linkage between bovine prolactin and BoLA-DRB3 genes: Genetics mapping
in cattle by single sperm typing. Genomics 13 : 44-48.
LIN, C.Y.; MC. ALLISTER, A.J.; NG-KWAI-HANG, K.F. and HAYES,
J.F. 1986. Effects of mik protein loci on fírst lactation production
in dairy cattle. Journal of Dairy Sciences 69:704-712.
LUM L. S., DOVC P. y J. F. Medrano. 1997. Polymorphisms of
bovine (-lactoglobulin promoter and differences in the binding aftinity of activator
protein-2 transcripton factor. J. Dairy Sci. 80: 1389-1397.
LUNDÉN , A., S. SIGURDARDÓTTIR, I. EDFORS-LILJA,
B. DANELL, J. RENDEL y L. ANDERSSON. 1990. The relationship between bovine major
histocompatibility complex class II polymorphism and disease studied by use
of bull breeding values. Animal Genetics 21 : 221-232.
MACHUGH, D. E., R. T. LOFTUS, D. G. BRADLEY, P. M. SHARP y
P. CUNNINGHAM. 1994. Microsatellite DNA variation within and among European
cattle breeds. Proc, R. Lond. B 256 :25-31.
MACHUGH, D. E., M. D. SHRIVER, R. T. LOFTUS, P. CUNNINGHAM
Y D. G. BRADLEY. 1997. Microsatellite DNA variation and the Evolution, Domestication
and Phylogeography of Taurine and Zebu Cattle. Genetics 146 : 1071 - 1086.
MCLEAN, DM, E.R.B. GRAHAM, R.W. PONZONI y H.A. MCKENZIE. 1984.
Effects of milk protein genetic variants on milk yield and composition. J. Dairy
Sci. 73 : 2647-2656.
MCLEAN, D.M. y J. SCHAAR. 1989. Effects of -lactoglobulin and
-casein genetic variants and concentration on syneresis of gels from renneted
heated milk. J. Dairy Research 56 : 296.
MEDJUGORAC, L; KUSTERMANN, W.; LAZAR, R; RUSS, I. and PIRCHNER,
F. 1994. Marker-derived phylogeny of European cattle supports demic expansion
of agriculture. Animal Genetics 25:19-27.
MEDRANO, J.F. y E. AGUILAR-CORDOBA. 1990. Polymerase chain
reaction amplification of bovine b-lactoglobulin genomic sequences and identification
of genetic variants by RFLP analysis. Biotechnology 1(1): 73-77.
MEJDELL, C.M., Æ
. LIE, H. SOLBU, E.F. ARNET y R.L. SPOONER. 1993. Associations of bovine lymphocyte
antigens with milk and meat production traits of Norwegian cattle. J. Dairy
Sci. 76: 3778 - 3784.
MEJDELL, C.M., H. SOLBU, R.L. SPOONER, E.F. ARNET y Æ.
LIE. 1994a. Changes in BoLA Class I alíele frequencies over years in
norwegian Breeding bulls, and the association of BoLA with perfomance traits
in young bulls. Acta Agric. Scand., Sect. A, Animal Sci. 44 : 73-78.
MEJDELL, CM, O. LIE, H. SOLBU, E.F. ARNET y R.L. SPOONER. 1994b.
Association of major histocompatibility complex antigens (BoLA-A) with AI bull
progeny test results for mastitis, ketosis and fertility in Norwegian cattle.
Animal Genetics 25(2) : 99 - 104.
NEI, M. 1987. Molecular evolutionary genetics. Columbia University
press. New York. pgs. 512.
NG-KWAI-HANG, K. F. , J. F. RAYES, J.E. MOXLEY y H. G. MONARDES.
1987. Variation in milk protein concentrations associated with genetic polymorphism
and enviromental factors. J. Dairy Sci. 70 : 563.
NG-KWAI-HANG, K. F, G. MONARDES y J.FRAYES. 1990.Associationbetween
genetic polymorphism of milk proteins and traits during three lactations. J.
Dairy Sci. 73 : 3414-3420.
NG-KWAI-HANG, K.F; RAYES, J.F.; MOXLEY, J.E. and MONARDES,
G. 1984. Association of genetic variants of casein and milk serum proteins with
milk, fat and ptotein production by dairy cattle. J. Dairy Sel. 67(4):835-840.
ÆSTERGÁRD,
H., B. KRISTENSEN y S. ANDERSEN. 1989. Investigacions in farm animals of associationss
between the MHC system and disease resistance and fertility. Livest. Prod. Sci.
22 : 49 - 67.
PARK CH., I. RUSS, Y. DA & H. A. LEWIN. 1995. Genetic mapping
of F13 A to BTA23 by sperm typing: Difference in recombination rate between
bulls in the DYA-PRL interval. Genomics 27:113-118.
PRIMO, A.T. 1992. El ganado bovino ibérico en las Américas:
Quinientos años después. Arch.Zootec. (extra):421-432.
RAYMOND M, ROUSSET F .1995. GENEPOP 1.2: a population genetics
sotfware for exact test and ecumenicism. J. Hered. 86, 248-249.
RIPOLI, M.V., G. GIOVAMBATTISTA, T.C. DE LUGA, F. LABARTA,
J. ECHENIQUE, S. CASAS, E. CARRIZO, M. SÁNCHEZ MERA y EN. DULOUT. 1999.
Formación de un plantel base de ganado bovino Criollo Argentino para
producción lechera. Efecto sobre las frecuencias génicas de los
loci de k-caseína, as1-caseína
y prolactina. Arch. Zootec. 48: (en prensa).
SAL PAZ, F.P. 1986. El ganado Criollo Argentino definiciones
y características. En Primera Jornada Nacional de Ganado Bovino Criollo
1:3-7.
SANCHEZ MERA, M.G. 1989. Avances en un proyecto Agrosilvopastoril.
Memorias X Reunión del Grupo Técnico Regional del Cono Sur en
Mejoramiento y Utilización de los Recursos Forrajeros del Área
Tropical y Subtropical. Talleres Gráficos INTA. Chile 460 B.A.. Pag.
21.
SCHOOK, L.B., H.A. LEWIN y D.G. MCLAREN. 1991. Gene mapping
techniques and applications. M. Dekker, inc. N. Y. USA. several production characteristics
in three Danish cattle breeds. Acta Agrie Scandll: 163-196.
SPELMAN, R.T., W. COPPIETERS, J.A.M. VAN ARENDONK y H. BOVENHUIS.
1996. Cuantitativo traits loci analysis for five milk production traits on chromosomes
six in the duch Holstein-Friesian population. Genetics (in press).
STEAR, M.J., T.S. POKORNY, N.E. MUGGLI y R.T. STONE. 1988.
The relationships of birth weight, preweaning gain and postweaning gain with
the bovine major histocompatibility system. J. Anim. Sci. 67 : 641 - 649.
TONG, P S., S. VINK, N. Y. FARKYE y J. F. MEDRANO. 1993. Effects
of genetic variants of mik protein on the yield of Cheddar cheese. Page 179
in Cheese Yield and factors Affecting Its Control. Int. Dairy. Fed., Cork, Ireland.
VAN DER POEL, J.J., M.A. GROENEN, R.LM. DLTKHOF, D. RUYTER
Y MJ. GIPHART 1990. Denucleotide sequence of the bovine MHC class II alpha gene:
DRA, DQA, and DYA. Inmunogenetics 31:29-36.
VAN EIJK M. J. T., J. A. STEWART-HAYNES & H. A. LEWIN.
1992. Extensive polymorphism of BoLA -DRB3 gene distinguished by PCR-RFLP Animal
Genetics 23: 483-496.
VELMALA, R.; MÄNTYSAARI, E.A. and MAKI-TANILA, A.. 1993.
Molecular genetic polymorphism at the -casein and -lactoglobulin loci in Finnish
dairy bulls. Agrie, Sel. Finí. 2:431-435.
VILKKI, H. J., D. J. de KONING, K. ELO, R. VELMALA, A. MAKI-TANILA.
1997. Multiple marker mapping of quantitative trait loci of Finnish dairy cattle
by regression. J. Dairy Sel. 80(1): 198-204.
WEIGEL, K.A., A.E. FREEMAN, M.E. KEHRLI JR, M.J. STEAR y D.H.
KELLEY (1990) Association of class I bovine lynphocyte antigen complex alleles
with health and production traits in dairy cattle. L Dairy Sel. 73 : 2538-2546.
WILKINS, R. L, H. W. DAVEY, T. T. WHEELEER y C. A. FORD. 1995.
Differential expression of b-lactoglobulin alleles A and B in dairy cattle.
Page 189 in Intracellular Signaling in the Mammary Gland. C. L Wilde, M. Peaker,
C. H. Knight, de. Plenum Press, New York, NY.
YANG, DA, D.S. ROGER, LA. STEWART y H. LERWIN. 1993. Milk and
fat yields decline in bovine leukemia virus-infected Holstein cattle with persisten!
lymphocytosis. Proc. Natl. Acad. Sel. USA. 90 : 6538-6541.
YAO J, S.E AGGREY, D. ZADWORNY, J.F. HAYES y U. KUHNLEIN. 1996a.
Sequence variations in the bovine growth hormone gene characterized by single-strand
conformation polymorphism (SSCP) analysis and their association with millk production
traits in Holsteins. Genetics 144: 1809- 1816.
YAO, J.; ZADWORNY, D; KUHNLEIN, U.; AGGREY, S.E. and HAYES,
J.F. 1996b. A Msp I polymorphism in the bovine ornithine decarboxylase gene
and its possible association with selection for milk production in Holstein
bulls. Animal Genetics 27:283-284.
ZANOTTI, M., M. LONGERI M., M. POLLI, G. CERIOTTI y G. POLI.
1995. BoLA class II polymorphism andimmune response to Mycobacterium bovis antigens
in vitro. J. Anim. Breed. Genet. 112: 391-400.
ZANOTTI, M., G. POLI, W. PONTI, M. POLLI, M. ROCCHI, E. BOLZANI,
M. LONGERI, S. RUSO, H.A. LEWIN y M.J.T. VAN EIJK. 1996. Association of BoLA
class II hplotypes with subclinical progression of bovine leukaemia virus infection
in Holstein-Friesian cattle. Animal Genetics 27 : 337-341.
ZAR J, H. 1984. Biostatistical Analysis. Pentice-Hall inc.
Englewood Cliffs, N.J., USA. pp. 718.
ZWIAUER, D. 1980. Beziehungen zwischen biochemischen Markergenen
und leistungseigenschaften bein Rind. Fortschr Tierz Züchtungsbiol 2. Parey,
Hamburg Berlin.