Arch. Med. Vet., Vol. XXXVI N°
2, 2004, pp. 119-136
REVISIONES BIBLIOGRAFICAS
Estructura, función e implicaciones
patológicas de las proteínas del Rubulavirus porcino
Proteins of Porcine
rubulavirus: Structure, function and pathological implications
G. SANTOS-LOPEZ 1,
Q.F.B., M. en C. Microbiol.; J. HERNANDEZ 3
M.V.Z., M. en C. Fisiol., Dr. en C. Vet.; M. T. BORRAZ-ARGÜELLO 1,
Biol.; H. RAMIREZ-MENDOZA 2 M.V.Z., M. en Prod.
Animal, Dr. en C. Vet.; V. VALLEJO 1, Biol., M.
en C. Bioméd., PhD; J. REYES-LEYVA 1*,
M.V.Z., M. en C. Bioméd., Dr. en C. Bioméd.
1 Laboratorio
de Virología, Centro de Investigación Biomédica de Oriente, Instituto Mexicano
del Seguro Social; Hospital General de Zona No. 5, Km. 4.5 Carretera Federal
Atlixco-Metepec; 74360 Metepec, Puebla, México.
2 Dept. de Nutrición Animal, Centro de Investigación
en Alimentación y Desarrollo, Hermosillo, Son, México.
3 Dept. Producción Animal Cerdos, Fac. de Medicina
Veterinaria y Zootecnia, UNAM.
* Correspondencia
Resumen
El Rubulavirus porcino
(RVP) es el agente etiológico de la enfermedad del ojo azul de los cerdos, entidad
patológica que se caracteriza por cuadros de meningoencefalitis y daño reproductor
en cerdos. Es un virus envuelto con genoma de ARN de polaridad negativa perteneciente
a la familia Paramyxoviridae. Posee una alta afinidad por el tejido
nervioso en cerdos neonatos y por el sistema reproductor en cerdos adultos.
Este virus posee seis proteínas estructurales y dos no estructurales. En los
paramixovirus algunas de estas proteínas promueven una mayor diseminación y
daño en los tejidos infectados, mientras que otras tienen actividades reguladoras,
manteniendo la integridad de los tejidos por más tiempo. En este trabajo se
describe la estructura y función de las proteínas del Rubulavirus porcino y
su papel en la patogenia viral.
Palabras clave:
Rubulavirus porcino, virus LPM, síndrome del ojo
azul, virus emergentes, meningoencefalitis, esterilidad.
Summary
Viral infections constitute serious
concerns for breeders and veterinarians world wide, so the knowledge of the
virus structure and biological properties is of primary importance in the design
of prophylactic and control strategies to combat viral diseases. The Porcine
rubulavirus (PoRV) is the causal agent of "blue eye" disease of swine,
an endemic disease in Mexico, that is characterized by fatal meningoencephalitis
in piglets and reproductive failures with loss of fertility in adult pigs. PoRV
is an enveloped virus with a negative single stranded RNA genome belonging to
the Paramyxoviridae family. This virus possesses six structural and
three non-structural proteins. Structural proteins play an important role in
the virus infectious cycle, promoting both virus dissemination and damage to
infected tissues, while some non-structural proteins play a regulatory role,
inducing viral persistence and preserving the tissues integrity to assure the
survival of virus progeny. The objectives of this review are 1) to analyze the
structure and function of paramyxoviral proteins and 2) to illustrate their
role on the pathological features of PRV infection.
Key words:
Porcine rubulavirus, LPMV, blue eye disease,
emerging viruses, meningoencephalitis, sterility.
INTRODUCCION
La enfermedad del ojo azul de
los cerdos es causada por el Rubulavirus porcino (RVP), también
denominado virus del síndrome del ojo azul (VSOA) o virus de La Piedad Michoacán
(VLPM). Esta enfermedad consiste en un complejo formado por alteraciones neurológicas,
reproductivas y respiratorias acompañadas por opacidad de la córnea (Moreno-López
y col., 1986; Stephano y col., 1988).
La enfermedad del ojo azul se
identificó inicialmente en granjas de La Piedad Michoacán, en 1980 (Stephano
y col., 1981, citado en Stephano y col., 1988), de ahí se
diseminó en pocos años a 16 estados del centro y noreste de la República Mexicana
(Fuentes y col., 1992); actualmente, el mayor impacto económico
se registra en los estados de Michoacán, Guanajuato y Jalisco, zona considerada
endémica (Stephano, 1999). La detección de anticuerpos antivirales
en bancos de sueros permitió identificar que el virus estaba presente en la
zona de La Piedad Michoacán por lo menos desde 1972, aunque no hay registros
de infecciones similares ocurridas en esas fechas (Rosales y
col., 1988). El origen del virus es incierto; análisis filogenéticos muestran
que el Rubulavirus porcino tiene un ancestro común a todos los paramixovirus,
pero excluyen la posibilidad de que este virus sea una variante de algún otro
rubulavirus (Berg y col., 1997; Svenda y col.,
1997, 2002).
Los signos clínicos varían de
acuerdo con la edad de los animales. En cerdos lactantes las manifestaciones
nerviosas se presentan en forma progresiva, aguda y generalmente son fatales
(Stephano y col., 1988). En los cerdos de 3 a 4 meses de
edad las manifestaciones neurológicas son escasas y la tasa de mortalidad es
baja. En cerdos adultos las lesiones se limitan al aparato reproductor: en las
hembras se presentan abortos, aumento de mortinatos y reducción de la fertilidad;
en machos se observa epididimitis, orquitis, atrofia testicular y una sensible
pérdida de fertilidad (Stephano y col., 1988; Campos
y Carvajal, 1989; Stephano, 1999; Ramírez-
Mendoza y col., 1997). La opacidad corneal se presenta solamente en 1 a
10% de los animales infectados de cualquier edad (Stephano y
col., 1988). En general, los parámetros productivos se ven gravemente afectados
y las pérdidas económicas son cuantiosas.
Infecciones experimentales en
cerdos machos adultos mostraron que el virus produce una inflamación severa
en la cabeza del epidídimo, destrucción del epitelio tubular y formación de
granulomas a partir del día 15 post-infección (p.i.). También se observó reducción
en la motilidad y concentración de espermatozoides. Entre los días 30-45 p.i.
se identificó la degeneración de túbulos seminíferos con infiltración linfocitaria
que condujo a atrofia testicular (Ramírez-Mendoza y col., 1997).
En otro estudio se infectaron
9 hembras a las 6 y 10 semanas de gestación. Ninguna de las hembras infectadas
mostró cambios clínicos a lo largo del experimento; sin embargo, todas desarrollaron
anticuerpos contra el virus a los 16 días p.i. Congestión local y hemorragias
de la placenta y endometrio fueron observadas en 2 hembras inoculadas a las
10 semanas de gestación. Los fetos disminuyeron de tamaño y presentaron hemorragias
difusas en la piel, deshidratación y autolisis. Fetos momificados fueron encontrados
en 6 de las 9 hembras infectadas. No se encontraron momias en las cerdas no
infectadas. El virus fue aislado del pulmón, tonsila y ovario de las hembras
infectadas, así como del cerebro, pulmón e hígado de los fetos (Hernández-
Jáuregui, 2003).
ESTRUCTURA VIRAL
Con base en sus características
biológicas y estructurales (Moreno-López y col., 1986; Stephano
y col., 1988) y por su similitud genómica con los virus de la parotiditis
humana (VPH), los virus de parainfluenza humana 2 y 4 y el virus 5 de simio
(Sundqvist y col., 1991; Berg y col., 1991,
1992) el virus de la enfermedad del ojo azul se clasificó
en el orden Mononegavirales, familia Paramyxoviridae, subfamilia Paramyxovirinae,
género Rubulavirus, especie Rubulavirus porcino (cuadro 1)
(Rima y col., 1995; Mayo, 2002). Este virus no presenta reacción
antigénica cruzada con los virus parainfluenza 1, 2, 3 y 4, con los virus de
la enfermedad de Newcastle, del sarampión, el sincicial respiratorio ni con
el virus de la parotiditis (Moreno-López y col., 1986; Stephano,
1999). Al microscopio electrónico se observan viriones pleomórficos, la
mayoría esféricos de 180 a 300 nm de diámetro, formados por una nucleocápside
helicoidal protegida por una envoltura lipoproteica (Moreno-López
y col., 1986), que presenta prolongaciones de 8-12 nm, que corresponden
a las glicoproteínas HN y F responsables de las actividades hemaglutinante,
hemolítica, de neuraminidasa y formadora de sincicios observadas en el Rubulavirus
porcino (Moreno-López y col., 1986; Reyes-Leyva
y col., 1999) (figura 1a).
CUADRO
1. Estructura
taxonómica del orden Mononegavirales |
Taxonomic structure
of order Mononegavirales |
| |
| Familia |
Subfamilia |
Género |
Virus de Interés |
| Paramyxoviridae |
Paramyxovirinae |
Respirovirus
Morbillivirus
Rubulavirus
Henipavirus
Avulavirus |
V. Sendai, Parainfluenza
(PI) 1, 3
V. Sarampión, Moquillo
V. Parotiditis, PI 2, 4, RVP
V. de Nipah, Hendra
V. de la enfermedad de Newcastle |
| |
Pneumovirinae |
Pneumovirus
Metapneumovirus |
V. sincitial respiratorio
V. de la rinotraqueitis del
pavo |
| Rhabdoviridae |
|
Vesiculovirus
Lyssavirus
Ephemerovirus
Cytorhabdovirus
Nucleorhabdovirus |
V. de la estomatitis
vesicular
V. de la rabia
V. de la fiebre efímera bovina
V. de la necrosis de la
lechuga
V. de la papa enana amarilla |
| Filoviridae |
|
Marburgvirus
Ebolavirus |
V. de Marburg
V. de Ebola |
| Bornaviridae |
|
Bornavirus |
V. de Borna |
| |
Modificado de Rima y col., 1995, y
Mayo, 2002.
Adapted from Rima et al., 1995
and Mayo, 2002. |
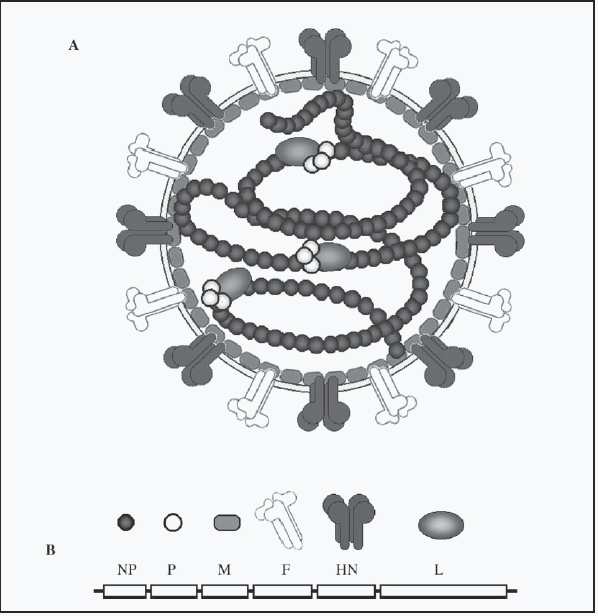 |
| |
FIGURA
1. a) Esquema del RVP y sus proteínas constituyentes. En la superficie
de la membrana viral se encuentran las glicoproteínas transmembranales
HN y F, en la parte interna de la misma se halla la proteína de matriz
M. Intimamente ligada a la cadena de ARN se encuentran las nucleoproteínas
(NP) que conforman la nucleocápside helicoidal y las proteínas P y L que
forman el complejo con actividad polimerasa del virus (Modificado de acuerdo
con Markwell y Fox, 1980; Rima y col.,
1995; Lamb y Kolakofsky, 1996; Hjertner,
1997; Reyes-Leyva y col., 2002); b) Mapa genómico
del RVP. El virus posee un genoma monocatenario de ARN negativo, formado
por seis genes separados por regiones intergénicas de longitud variable
(1 - 47 nucleótidos) (Linné y col., 1992). |
a) PoRV and its proteins.
In the virus membrane (envelope) are inserted the transmembrane HN and
F glycoproteins, on the inner face is located the matriz (M) protein,
which maintains an intimate contact among the nucleocapsid and glycoproteins.
The genomic RNA is protected by the nucleoprotein (NP) and have an helical
structure, which is also associated with the P and L proteins. These last
proteins constitute the viral polymerase complex (Adapted from Markwell
and Fox, 1980; Rima et al., 1995;
Lamb and Kolakofsky, 1996; Hjertner,
1997; Reyes-Leyva et al., 2002); b) Genomic map
of PoRV. This virus possesses a negative single stranded RNA genome which
contain six genes separated by variable length intergenic regions (1-47
nucleotides) (Linné et al., 1992). |
La información genética del RVP
está contenida en una molécula de ácido ribonucleico (ARN) de cadena negativa,
es decir, su orientación es contraria a la del ARN mensajero. A partir de esta
molécula se sintetiza una cadena de ARN de sentido positivo que actúa como molde
para la replicación. El virus posee 6 genes que codifican 10 proteínas con funciones
estructurales, reguladoras y enzimáticas. La organización del genoma es la siguiente:
3' NP-P-M-FHN- L 5' (figura 1b); esta secuencia y las regiones
intergénicas (segmentos de 1 a 47 nucleótidos) son similares en todos los miembros
del género Rubulavirus (Linné y col., 1992; Lamb
y Kolakofsky, 1996). Cada gen codifica una proteína, excepto el gen F, que
codifica el polipéptido F0 que es dividido por una proteasa celular en las proteínas
F1 y F2 (Berg y col., 1997), y el gen P que codifica las proteínas
P, I, V y C, que se expresan por edición del ARN mensajero (Berg
y col., 1992) (cuadro 2).
CUADRO
2. Genes y proteínas del Rubulavirus porcino |
Genes and proteins
of Porcine rubulavirus |
| |
Gen |
Péptido |
Nombre |
Masa
molecular |
Características,
ubicación y actividades relacionadas |
NP |
NP |
Nucleoproteína |
68 |
Proteína de
la nucleocápside. Función estructural, protección
del genoma y conformación tridimensional del substrato de la polimerasa. |
P |
P |
|
52 |
Asociada a la nucleocápside.
Forma el complejo polimerasa con la proteína L. |
V |
V |
. |
N.D |
Proteína no
estructural. Actividad inhibitoria sobre el complejo
polimerasa viral. Inhibición de la cascada de señales activada
por INFy |
C |
C |
|
N.D |
Proteína no
estructural. Actividad inhibitoria sobre el complejo
polimerasa viral. Inhibición de la cascada de señales activada
por INFy |
I |
I |
|
N.D |
Proteína no
estructural. Función desconocida. |
M |
M |
Proteína de Matriz |
40 |
Proteína asociada a
la membrana. Participa en el ensamble de los viriones. |
F |
F1 |
Proteína de Fusión |
59 |
Glicoproteína
transmembranal. Péptido de fusión de membranas
viral y celular, hemólisis, formación de sincitios. |
|
F2 |
|
12 |
Glicoproteína
externa de membrana. Asociada a F2 mediante
enlace disulfuro. |
HN |
HN |
Hemaglutinina-
Neuraminidasa |
66 |
Glicoproteína
transmembranal. Reconocimiento del receptor celular y adherencia. Hidrólisis
de ácido siálico, promoción de la fusión membranal
célula-virus. |
L |
L |
Proteína de alto
peso molecular |
200 |
Enzima asociada a la
nucleocápside. Componente catalítico del complejo polimerasa
formado con la proteína P. Transcripción y replicación. |
El RVP conserva de 41-47% de
identidad en secuencia genética con el virus de la parotiditis humana (Sundqvist
y col., 1991; Berg y col., 1991, 1992,
1997; Svenda y col., 1997); esto influye
para que ambos virus presenten similitudes en actividades biológicas y en características
patológicas, pero las similitudes más importantes se refieren a su afinidad
y tropismo tisular. Así estos dos virus comparten su afinidad por el sistema
nervioso central, por órganos del aparato reproductor femenino y masculino,
por tejido glandular y por células y tejidos linfáticos (Stephano
y col., 1988; Allan y col., 1996; Ramírez-Mendoza
y col., 1997; Ramírez-Herrera y col., 1997; Hernández
y col., 2002; Wolinsky, 1996).
PROTEINAS DE LA ENVOLTURA
VIRAL
La membrana del RVP presenta
tres tipos de proteínas: las glicoproteínas HN y F son de transmembrana con
dominios activos en el exterior del virión y la proteína M asociada a la parte
interna de la membrana.
La glicoproteína HN de los paramixovirus
es responsable de reconocer el receptor y de la adhesión a las células blanco
de la infección. La HN es una proteína multifuncional que presenta actividad
hemaglutinante, neuraminidasa y promotora de fusión de las membranas viral y
celular (Ebata y col., 1991; Sergel y col.,
1993; Lamb, 1993). Esta proteína forma dímeros y tetrámeros
que exponen varios sitios de unión al receptor celular sobre la membrana viral
(Markwell y Fox, 1980, Reyes-Leyva y col.,
2002). El análisis del ADN complementario del gen HN del RVP mostró que
codifica una proteína de 576 aminoácidos, con un dominio hidrofóbico cercano
a la región amino terminal, por la que se ancla a la membrana viral, y con cuatro
sitios potenciales de N-glicosilación (Sundqvist y col., 1991).
El análisis de la glicoproteína
HN, purificada mediante centrifugación diferencial en gradientes de sacarosa
y cromatografía de intercambio iónico, permitió determinar que está compuesta
por 50.5, 13.3 y 13.6% de aminoácidos no polares, polares sin carga y polares
cargados. Su fracción glicosídica está constituida por manosa, galactosa, N-acetilgalactosamina
y N-acetilglucosamina en proporciones de 3:3:2:1 respectivamente, así como trazas
de ácido N-acetilneuramínico (Reyes-Leyva y col., 1999).
Lo que muestra la proporción y los constituyentes típicos de una proteína N-glicosilada,
coincidiendo con las predicciones de composición peptídica y de N-glicosilación
basadas en la secuencia genética (Sundqvist y col., 1991;
Zenteno-Cuevas y col., 1998). No obstante, la proporción
encontrada de N-acetilgalactosamina sugiere que esta proteína también podría
estar O-glicosilada.
En relación a su actividad, la
glicoproteína HN aglutina eritrocitos de una gran variedad de especies animales,
incluyendo cerdo, carnero, vaca, perro, conejo, ratón, rata, hámster, cobayo,
pollo y humano tipos A, B, y O (Stephano y col., 1988; Moreno-López
y col., 1986; Reyes-Leyva y col., 1993). Esto se debe
al reconocimiento específico de ácidos siálicos expresados en las superficies
de todos estos eritrocitos. Al respecto, hemos demostrado, en ensayos de competencia
con diversos azúcares, que la proteína HN posee mayor afinidad por residuos
de ácido N-acetilneuramínico unidos por enlace glicosídico alfa 2-3 a galactosa
(NeuAca2,3Gal), los cuales inhiben su actividad hemaglutinante (Reyes-
Leyva y col., 1993, 1999). La proteína HN posee también actividad neuraminidasa
o sialidasa que le permite hidrolizar los residuos de ácido siálico de los receptores
celulares; esta actividad se realiza a un pH de 3.3 y es muy estable en un rango
de temperatura de 30-55°C (Santos-López y col., 2004).
La expresión de glicoconjugados
con NeuAca2,3Gal es determinante de la susceptibilidad a la infección por el
RVP en cultivos celulares. La competencia por el receptor con lectinas (proteínas
que reconocen carbohidratos) específicas para este oligosacárido terminal, así
como inhibidores de la glicosilación como la dexametasona, que favorece la expresión
de ácido neuramínico en enlace a2.6 en lugar de a2.3, lograron bloquear la unión
del virus al receptor celular y evitar así la infección, confirmando la importancia
del ácido siálico en posición a2.3 para que la infección se lleve a cabo exitosamente
(Reyes-Leyva y col., 1997).
Hemos identificado una estrecha
relación entre la expresión tisular de NeuAca2,3Gal en cerdos sanos (Vallejo
y col., 2000) con los sitios de replicación viral identificados en cerdos
infectados natural y experimentalmente (Allan y col., 1996;
McNeilly y col., 1997; Ramírez-Mendoza y col.,
1997). Los datos indican que las diferencias patológicas entre distintos
grupos de edad se deben en parte a la expresión diferencial de NeuAca2,3Gal
en los tejidos, ya que se encuentra una mayor proporción de este oligosacárido
en el sistema nervioso central de cerdos neonatos y en el sistema reproductor
de cerdos adultos que en otros tejidos. Las diferencias en la expresión tisular
de ácidos siálicos parecen guardar una relación con cambios hormonales durante
el proceso de maduración de los animales (Vallejo y col., 2000;
Reyes-Leyva y col., 2002).
La glicoproteína F es muy conservada
entre los miembros de la familia Paramyxoviridae y otras familias de
virus formados por ARN de cadena negativa. Esta proteína tiene un dominio altamente
hidrofóbico que participa en la fusión de la membrana celular y la envoltura
viral, lo que permite que el virus se introduzca al citoplasma y que se difunda
de célula a célula sin exponerse al medio extracelular (figura
2). Esta característica origina la formación de células gigantes
multinucleadas (sincicios) apreciables en tejidos y en cultivos celulares infectados
por paramixovirus (Morrison y Portner, 1991). La proteína
F es sintetizada como un precursor inactivo llamado F0, que por acción
de una proteasa celular del tipo de la tripsina da origen a dos polipéptidos
(F1 y F2) unidos por un enlace disulfuro que
constituyen la proteína activa (Sheid y Choppin, 1974). El
procesamiento proteolítico expone un segmento altamente hidrofóbico en el extremo
N-terminal de la subunidad F1, implicado directamente en el ataque
inicial a la membrana celular para dar lugar a la fusión (White,
1990).
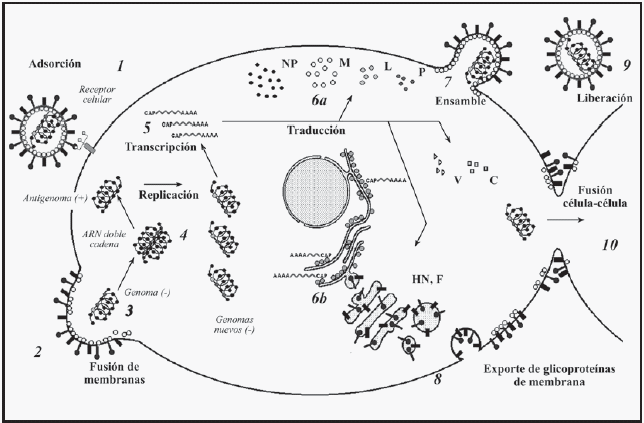 |
| |
FIGURA
2. Representación gráfica del ciclo de los paramixovirus. La
partícula viral se adsorbe en la membrana de la célula hospedera por medio
de la proteína HN, que reconoce como receptor específico el oligosacárido
NeuAca2,3Gal (1). Un cambio conformacional de la HN activa la proteína
F, la cual lleva a cabo la fusión de las membranas celular y viral (2),
provocando que el ARN viral sea liberado en el citoplasma (3). En este
punto comienza, por un lado, la síntesis de ARN antigenómico (cadena complementaria
al genoma), la cual da origen a genomas nuevos (4), y por otro lado, a
la producción de ARN mensajero (5), que codifica para las proteínas virales
(6a). Los productos de la traducción se dirigen al sitio de ensamble,
las proteínas NP, P y L son acopladas al ARN recién sintetizado y la proteína
M se ubica en la parte interna de la membrana celular (7). Las glicoproteínas
(HN y F), sintetizadas en retículo endoplásmico, son modificadas en aparato
de Golgi (6b) y posteriormente expresadas en la membrana citoplásmica,
en contacto íntimo con la proteína M (8). La afinidad de las proteínas
del genoma (NP, P y L) con la proteína de matriz y de ésta con las glicoproteínas
es determinante para el ensamble del virión, que es liberado de la célula
por exocitosis (9). Es característico en los paramixovirus que además
de la formación de viriones, los virus pueden infectar las células inmediatamente
vecinas a través de la fusión membranal célula-célula, debido a la expresión
de las proteínas virales en la membrana de la célula hospedera (10), lo
cual le permite dispersarse sin necesidad de salir al medio extracelular
(Modificado de Lamb y Kolakofsky, 1996, y Reyes-Leyva
y col., 2002). |
Graphic representation
of the cycle of paramyxoviruses. The virus particle is adsorbed on the
cellular membrane by means of the HN protein, which recognizes as specific
receptor the oligosacharide NeuAca2,3Gal (1). A conformational change
in HN seems to cause the activation of the F protein and then the membrane
fusion is carried out (2) internalizing the virus genome in the cytoplasm
(3). Once the genome is inside the cell, it begins the synthesis of antigenomic
RNA (a positive + complementary copy of the genome) which serves as an
intermediate in the genomic RNA (-) replication (4). Messenger RNAs for
each protein are synthesized from genomic RNA (5). The translation products
become either soluble (6a) or endoplasmic reticulum associated (glycoproteins)
(6b), they are transported to the assembling sites (7), where the M protein
plays a decisive role in the assembly of virions, by electrostatic interactions
with the transmembrane glycoproteins and nucleocapsid (8). Later, virions
are released by exocytosis (9). Additionally, the virus particles can
be disseminated to neighboring cells by means of cell-cell membrane fusion,
due to the expression of glycoproteins on infected cell membrane and receptors
in the others (10) in this form viruses avoid exposition to unfavorable
environment (Adapted from Lamb and Kolakofsky, 1996
and Reyes-Leyva et al., 2002). |
Se ha propuesto que la proteína
F debe ser activada de alguna forma por la HN para que el evento de fusión de
membranas se desencadene. En estos eventos están relacionados dominios cercanos
a la membrana viral, donde se encuentran motivos de unión proteína-proteína
(Deng y col., 1995; Hu y col., 1992; Stone-Hulslander
y Morrison, 1999; Tsurudome y col., 1995). Al parecer,
durante la interacción con los receptores celulares la HN sufre un cambio de
conformación que permite contactar un dominio de la proteína F, que a su vez
expone el péptido hidrofóbico involucrado directamente en el ataque a la membrana
celular, para iniciar de esta forma la introducción a la célula hospedera. Esta
interacción F-HN es específica de especie viral, ya que se ha observado que
la coexpresión en cultivos celulares de proteínas de distintos paramixovirus
no producen fusión celular (Deng y col., 1995).
La proteína M se localiza en
la parte interna de la envoltura viral, está constituida por 369 aminoácidos,
gran parte de los cuales son de carácter básico (Berg y col.,
1992). Debido a su carga neta positiva tiene gran afinidad por la NP, característica
importante que le permite intervenir en el ensamble de la partícula viral con
un reducido gasto energético, interactuando con las nucleocápsides en las zonas
de la membrana celular donde se están expresando las glicoproteínas virales
(Sanderson y col., 1993). La proteína M es importante en
el mantenimiento de la estructura del virión, ya que forma una especie de puente
entre la nucleocápside y la envoltura viral; además, se ha propuesto que juega
un papel regulador de la transcripción, probablemente por obstaculizar la acción
de la polimerasa sobre el genoma viral durante el ensamble de viriones (Lamb
y Kolakofsky, 1996).
PROTEINAS ASOCIADAS A
LA NUCLEOCAPSIDE
La nucleoproteína (NP) forma
parte integral de la nucleocápside y rodea al ARN genómico en todo su contorno
helicoidal. Tiene funciónroporciona una conformación adecuada a la cadena de
ARN genómico durante su transcripción y replicación (Kolakofsky
y col., 1998). Cada molécula de NP está asociada a seis nucleótidos y se
propone que ésta es la razón por la que solamente los genomas paramixovirales,
que poseen un número de nucleótidos múltiplo de seis, son replicados eficientemente
(Calain y Roux, 1993; Kolakofsky y col., 1998).
La unión de la NP al genoma es de una afinidad muy alta y no se separan durante
la replicación ni la transcripción, así que, a diferencia de los virus de ARN
positivo, el genoma de los virus de ARN negativo nunca se encuentra desnudo
(Kolakofsky y col., 1998).
A la nucleocápside se asocian
aproximadamente cincuenta complejos con actividad de polimerasa que se encargan
de la transcripción y replicación del genoma viral. Cada complejo está formado
por tres unidades de la proteína P y una de la proteína L, aunque también se
puede presentar el mismo número de complejos de proteína P sin la proteína L
(Kolakofsky y col., 1998). El gen P puede dar origen a cuatro
productos: P, V, I y C. La proteína V, de 249 aminoácidos, es codificada por
una copia exacta del gen P. La adición de dos guanidinas no codificadas, entre
las posiciones 586 a 591 de la secuencia genómica, permite que el marco de lectura
se alargue para dar lugar a la expresión de la proteína P, de 404 aminoácidos.
El marco de lectura que da origen a la proteína I se forma de manera similar
al de la proteína P, sólo que en lugar de añadir dos guanidinas se añade una,
lo que ocasiona que el marco de lectura termine antes que cualquiera de los
otros dos, originando un péptido de 174 aminoácidos. Por último, la proteína
C (126 aminoácidos) se codifica en un marco de lectura más pequeño contenido
dentro de los tres anteriores (figura 3).
De esta manera se puede observar que las proteínas V, P e I poseen un dominio
idéntico en el extremo amino de 168 aminoácidos (Berg y col.,
1992).
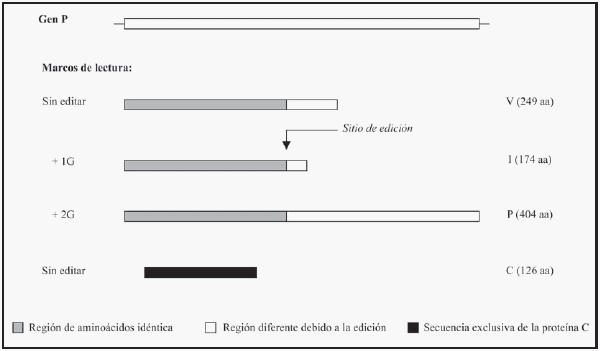 |
| |
FIGURA
3. Edición cotranscripcional del gen P. Los paramixovirus poseen un característico
proceso de edición en el gen P, que consiste en la adición de una o dos
guanidinas (G) que no se encuentran codificadas en la secuencia génica.
La adición de estos nucleótidos ocurre en un sitio con la secuencia UUUAAGAGGGGGG,
conservado en muchos miembros de la familia y que en el caso del RVP se
sitúa entre los nucleótidos 579 a 591. La inserción de los nucleótidos
se lleva a cabo en cualquier posición entre las últimas G de la secuencia
mencionada, por lo que el proceso es denominado "tartamudeo" de la polimerasa.
Originalmente existe un marco de lectura que codifica la proteínas V.
Cuando es agregada 1 G, este marco de lectura se acorta para dar origen
a la proteína I, mientras que al agregarse 2 G, el codón de terminación
se desplaza produciendo un marco de lectura más largo que da origen a
la proteína P. La proteína C puede ser traducida en cualquiera de los
mensajeros, ya sea editados o no, porque su marco de lectura se encuentra
en cualquiera de ellos y no es modificado por el proceso de edición (Berg
y col., 1992; Linné y col., 1992). |
Co-transcriptional
edition of the P gene. PoRV, like other Paramyxoviridae members, possesses
a typical edition process in the P gene, which consist in the addition
of 1 or 2 G nontemplated nucleotides. This nucleotides are added in the
579-591 site of P gene, where is located the sequence UUUAAGAGGGGGG conserved
in all the viruses of the family. Nontemplated G residues ares inserted
between the others G's in the sequence, in a process so-called "polymerase
stuttering". In the nonedited P gene there is one open reading frame (ORF)
that codes for the V protein. When 1 G is added this ORF is shortened
to originate the I protein mRNA; however, when 2 G are added the end codon
is displaced far from, producing a longer ORF which originates the P protein.
The C protein can be translated by anyone of these mRNA, since its ORF
is present in all they and the edition process don't modify its sequence
(Berg et al., 1992; Linné et
al., 1992). |
Datos experimentales muestran
que las proteínas V y C de los paramixovirus están involucradas en la regulación
de la replicación y la transcripción viral (Curran y col., 1991
y 1992; Kato y col., 1997; Hjertner
y col., 1998a y b) y en la inhibición de las cascadas
de activación del interferón en la célula hospedera como respuesta a la infección
(Didcock y col., 1999a, b). Estas evidencias
se discuten más adelante en esta revisión.
La proteína L es la más voluminosa
del virión y es el componente catalítico de la ARN polimerasa (Svenda
y col., 1997). La proteína L del RVP, y de los paramixovirus en general,
guarda gran homología en función con la proteína L del Virus de
la estomatitis vesicular (VSV), miembro de la familia
Rhabdoviridae (Svenda y col., 1997). Las funciones
de la proteína L del VSV se han estudiado intensamente, entre ellas se encuentran
actividades catalíticas en la síntesis de ARN genómico y mensajero, actividades
de metiltransferasa (Hercyk y col., 1988), de cinasa (Sánchez
y col., 1985) y de poliadenil sintetasa (Hunt y Hutchinson,
1993). La gran similitud en secuencia genética entre la proteína L del VSV
y la de los paramixovirus sugiere que estas proteínas podrían tener las mismas
funciones. Estas actividades explican por qué los paramixovirus realizan sus
ciclos completos de replicación en citoplasma sin necesidad de ingresar al núcleo,
ya que la proteína L proveería de actividades de replicación del genoma completo
para dar origen a nuevas partículas virales, síntesis de ARN mensajero para
expresar sus genes, así como modificación de los mismos: metilación en su extremo
5' y poliadenilación en el extremo 3' para que estos funcionen en forma similar
a los ARN mensajeros celulares.
Recientemente se demostró la
interacción de las proteínas V y P con la NP y la proteína L del Rubulavirus
porcino. En estos ensayos se observó que la unión de V y P a la NP
se lleva a cabo por medio de su extremo carboxilo terminal y que el extremo
amino, que es común en ambas proteínas, es el que interacciona directamente
con la proteína L (Svenda y col., 2002). Sin embargo, poco
se sabe del papel que juegan los complejos P-L y V-L en los procesos de transcripción
y replicación viral. Se ha propuesto que P es un factor que promueve la transcripción
y que V actúa como regulador (Lamb y Kolakofsky, 1996).
TROPISMO Y SUSCEPTIBILIDAD
TISULAR
Diversos reportes de infecciones
experimentales con el RVP coinciden en que el aislamiento y la determinación
de antígeno viral en los cerdos infectados son esencialmente en sistema nervioso
central de neonatos (Stephano y col., 1988; Allan
y col., 1996; McNeilly y col., 1997; Ramírez-Herrera
y col., 1997) y en aparato reproductor de cerdos adultos (Stephano
y col., 1988; Campos y Carvajal., 1989; Ramírez-Mendoza
y col., 1997; Reyes-Leyva y col., 2002). La infección
experimental con la cepa LPM del Rubulavirus porcino en cerdos de 3 días de
edad produjo signos neurológicos severos, muriendo a los 8 días p.i. En contraste,
los cerdos de 17 días mostraron cierta resistencia y solamente el 30% de ellos
presentó signos nerviosos (Allan y col., 1996). Las infecciones
experimentales en cerdos de más de 20 días de edad con la cepa LPM no han mostrado
la severidad de los cuadros neurológicos observados en los animales más jóvenes,
o definitivamente no han mostrado ningún signo clínico (Moreno-López
y col., 1986; Allan y col., 1996; Ramírez-Mendoza datos
no publicados). La distribución del antígeno viral detectado por inmunofluorescencia
en los tejidos de los animales infectados por vía intranasal sugiere que el
RVP ingresa por el nervio olfatorio hacia el sistema nervioso central (Allan
y col., 1996; Ramírez-Herrera y col., 1997). Estos resultados
correlacionan directamente con la distribución de la molécula reconocida en
forma específica por la proteína HN (NeuAca2,3Gal) en los cerdos neonatos y
lactantes, en los que el nervio olfatorio expresa una gran cantidad de este
carbohidrato (Vallejo y col., 2000), lo cual refuerza la
hipótesis de que en forma natural la entrada del virus es por vía nasal (Hernández-
López y col., 1997).
La expresión de NeuAcα2,3Gal
parece ser determinante también de la susceptibilidad a la infección en el aparato
reproductor de cerdos adultos (Vallejo y col., 2000). Durante
una infección experimental con la cepa PAC3 (Jalisco/ 1993) en cerdos machos
adultos se logró detectar al antígeno viral en el epidídimo, testículos y próstata
hasta por 70 días p.i., lo que sugiere que el virus puede ser excretado con
el semen y que podría transmitirse la enfermedad del ojo azul por vía venérea
(Ramírez-Mendoza y col., 1997).
Recientemente se detectó la presencia
transitoria del virus en células sanguíneas de cerdos infectados experimentalmente,
en la primera semana p.i., asociado a la superficie de eritrocitos y posteriormente
localizado en el interior de leucocitos (Reyes-Leyva y col.,
2004). Esto último coincide con la identificación de antígeno viral en el
interior de monocitos infiltrados en el epidídimo de cerdos infectados a las
7 semanas p.i. (Ramírez-Mendoza y col., 1997). Se ha demostrado
que la infección de células inmunes juega un papel importante en la diseminación
sistémica de otros paramixovirus (Katz, 1994; Wolinsky,
1996).
PAPEL DE LAS PROTEINAS
VIRALES EN LA PERSISTENCIA VIRAL
La persistencia se refiere a
la presencia de partículas virales en un organismo o tejido durante períodos
relativamente largos, comparados con el tiempo que permanecen durante una infección
lítica aguda. Durante todo este tiempo los virus presentan un bajo nivel de
replicación y la infección puede pasar clínicamente desapercibida. Sin embargo,
en algún momento la población viral puede volver a desencadenar una infección
lítica, debido a múltiples factores. Tanto en un estado como en otro, un elemento
muy importante es la baja expresión o mal funcionamiento de algunas proteínas
virales.
Recientemente se demostró la
persistencia del RVP en el sistema nervioso central de cerdos convalecientes
a la infección natural (Cuevas- Romero, 2002) y experimental
(Wiman y col., 1998). En este fenómeno de persistencia, que
parece ser común entre los paramixovirus, se han involucrado distintos mecanismos
como la producción de partículas virales defectuosas, que no poseen un genoma
completo y no son replicadas eficientemente (Lazzarini y col.,
1981; Murphy y col., 1987), por lo que compiten con el
genoma viral completo, por la polimerasa, evitando así que la replicación viral
alcance un nivel óptimo (Hjertner, 1997). Se ha observado
que la expresión de proteínas virales funcionalmente deficientes son importantes
para el establecimiento de una infección persistente; por ejemplo, una proteína
F defectuosa es responsable de disminuir los patrones de fusión celular durante
la infección por virus del sarampión (Hummel y col., 1994).
En otro estudio con el virus del sarampión se reportó que una cepa persistente
reduce la expresión de las proteínas P, M y H (Celma y Fernández-
Muñoz, 1992). Otros trabajos involucran a la proteína M, que muestra disfunciones
en virus aislados de pacientes con panencefalitis esclerosante subaguda (Rima
y col., 1994), y a la proteína V, cuya ausencia en cultivos celulares aumenta
la virulencia del virus Sendai (Kato y col., 1997). Por otro
lado, cuando la HN incrementa su actividad de neuraminidasa promueve la persistencia
del virus parainfluenza 3 en cultivos celulares. Observaciones realizadas en
virus con una actividad de neuraminidasa alta sugieren que la proteína HN expresada
en la membrana de células infectadas hidroliza el ácido siálico de las células
vecinas, reduciendo la cantidad necesaria de receptores para que el virus pueda
introducirse a la otra célula en forma efectiva (Moscona y Pelusso,
1991).
La falta de respuesta inmune
citotóxica también se ha involucrado en el desarrollo de infecciones persistentes
(Rima y col., 1994; Murphy, 1994); un
fenómeno importante para este efecto es la reducción en los niveles de expresión
de las proteínas virales expresadas en la superficie de las células hospederas
(Smith, 1994).
La inducción de la persistencia
in vitro del RVP se ha llevado a cabo en células PK-15, mostrando
la presencia de ARN subgenómico y una gran disminución en el transcrito del
gen L, componente catalítico de la polimerasa. Se observó también un ligero
descenso del ARN mensajero de la proteína P, acompañado por elevación de la
proteína V (Hjertner y col., 1998a y b).
La proteína V ha sido relacionada con la regulación negativa de la transcripción
y replicación, originando que los virus sean menos nocivos para las células
(Curran y col., 1991; Kato y col., 1997).
In vivo el RVP fue capaz de permanecer en cerdos convalecientes a una infección
experimental por períodos relativamente largos. El ARN viral pudo detectarse
en cerebro y pulmón mediante ensayos de hibridación con sondas de ARN, después
que los cerdos convalecientes fueron inmunosuprimidos (Wiman
y col., 1998). Tales hallazgos sugieren que el RVP, como la mayoría de los
paramixovirus, es capaz de permanecer por períodos prolongados produciendo una
infección persistente subclínica en el hospedero; este hecho puede tener un
peso epidemiológico importante en la enfermedad, ya que un animal infectado
asintomático puede servir como reservorio y fuente de infección para otros cerdos.
La importancia de la persistencia viral en la diseminación de la enfermedad
ha sido confirmada recientemente en cerdos convalecientes a la infección natural
que transmitieron el virus a cerdos centinelas durante la fase subclínica de
la enfermedad (Cuevas-Romero, 2002).
ASPECTOS INMUNOLOGICOS
Con relación a la respuesta inmune
humoral, se ha demostrado la producción de anticuerpos en los cerdos infectados
naturalmente, los cuales pueden permanecer por lo menos durante año y medio
en los animales que sobreviven (Stephano, 1999). En cerdos
infectados experimentalmente nuestro grupo ha observado que la producción de
anticuerpos se identifica por ensayos de neutralización viral e inhibición de
la hemaglutinación a partir de la primera y segunda semanas p.i., respectivamente
(Hernández y col., 1998). La aplicación de una vacuna constituida
por virus inactivado con formalina en hembras sanas durante el segundo tercio
de la gestación indujo anticuerpos que fueron transferidos a la camada y fueron
detectables en los lechones durante los 30 días posteriores al nacimiento. La
aplicación de esta vacuna experimental en una granja afectada por la enfermedad
del ojo azul indujo una notable producción de anticuerpos; sin embargo, sólo
redujo un 30% las manifestaciones clínicas y la mortalidad por la infección
(Hernández- Jáuregui y col., 1992), lo cual sugiere que existen
otros mecanismos que están involucrados en el control de la enfermedad.
Por su ubicación en la superficie
y su complejidad, la proteína de mayor potencial antigénico y el blanco más
efectivo del sistema inmunológico es la HN. Por ello esta proteína es también
la que presenta mayor variabilidad en muchos de los virus de esta familia y
también la que más se estudia como candidato en la generación de vacunas (Ray
y Compans, 1990; Haanes y col., 1997; Yates
y col., 1997). Nosotros hemos demostrado que la proteína HN del RVP es el
antígeno inmunodominante en la respuesta inmune de cerdos infectados experimentalmente.
Los anticuerpos anti-HN se detectaron por inmunotransferencia a los 15 días
p.i.; mientras que la producción de anticuerpos contra los antígenos NP y M
del virión se iniciaron a las cuarta y quinta semanas p.i., respectivamente
(Hernández y col., 1998).
Otro factor importante de protección
contra paramixovirus es la glicoproteína F, ya que se ha observado que en los
virus de parainfluenza el desarrollo de anticuerpos contra F y HN brinda mejor
protección que cualquiera de las dos por separado; sin embargo, la inmunogenicidad
de la proteína F es bastante baja, por lo que es necesario repetir varias veces
la inoculación de la misma para obtener resultados satisfactorios (Ray
y Compans, 1990).
En relación a la respuesta inmune
celular, se ha demostrado que las células mononucleares de cerdos infectados
responden proliferando al estímulo antigénico con el RVP in vitro. Esta respuesta
proliferativa se presentó a partir de la segunda semana p.i. y se mantuvo al
menos durante 7 semanas, alcanzando el punto máximo a la cuarta semana p.i.
Las células que proliferan en respuesta al virus son principalmente linfocitos
T con fenotipo CD4+CD8+ y CD4+CD8-. Esos resultados sugieren que la estimulación
antigénica con el RVP activa vías de señalización de los linfocitos diferentes
a las utilizadas por otros antígenos y mitógenos (Hernández y
col., 1998, 2001, 2004).
La respuesta celular tiene un
peso muy importante para la erradicación de la infección viral. Se ha observado
que las glicoproteínas HN y F de diversos paramixovirus pueden estimular células
T citotóxicas y NK, pero la respuesta celular más efectiva es la dirigida contra
antígenos de la estructura interna del virus, como la proteína NP, cuyos péptidos,
presentados por las moléculas del complejo principal de histocompatibilidad
tipo I, inducen la lisis de las células infectadas (Ray y Compans,
1990).
EVASION DE LA RESPUESTA
INMUNE
En años recientes se ha investigado
el papel de ciertas proteínas de los paramixovirus que participan en la inhibición
de la actividad del interferón (IFN). Los interferones son una familia de proteínas
que se secretan en respuesta a infecciones virales. Esencialmente son de dos
tipos, los INF>α/ß,
producidos en respuesta directa a la infección viral por fibroblastos y diversos
tipos celulares, y el IFNγ,
producido por linfocitos T y células NK. Los interferones tienen receptores
específicos en las membranas de las células, cuyo reconocimiento activa la transcripción
de diversos genes involucrados en actividades antivirales. Cuando el IFN reconoce
su receptor en una célula se activa una cascada de señales que propicia la fosforilación
de los factores de transcripción STAT1 y STAT2, los cuales se dimerizan y se
internan en el núcleo, donde interaccionan con la proteína p48 para formar un
complejo de activación transcripcional de genes que responden a INFα/ß
(Stark y col., 1998; Goodbourn y col., 2000).
Se ha observado la interacción de las proteínas celulares STAT con las proteínas
virales V y C durante infecciones por los paramixovirus Sendai (Didcock
y col., 1999b), SV5 (Didcock y col., 1999a, b)
y el de parotiditis (Kubota y col., 2001; Nishio
y col., 2002). En cada caso se ha comprobado que esta interacción ocasiona
la disminución de los niveles de las proteínas STAT, lo cual interrumpe las
vías de señalización del interferón y se inactiva la respuesta antiviral. En
el caso del virus SV5 se logró probar que la unión de su proteína V a STAT1
promueve la degradación proteolítica de ésta (Didcock y col.,
1999a), mientras que en un estudio con el virus de parotiditis se observó
que su proteína V se une tanto a STAT1 como a STAT2 (Nishio y
col., 2002). En este último trabajo se demostró también la interacción de
la proteína C del virus Sendai con STAT1, por lo que son dos las proteínas de
este virus que pueden unirse a STAT1, sin embargo, no se sabe si existe un efecto
de competencia entre ambas o bien un fenómeno de sinergismo. Un estudio con
el virus Nipah, otro miembro de la familia Paramyxoviridae que afecta
también al ganado porcino, muestra que su proteína V interacciona con las proteínas
STAT1 y 2, pero que no induce su degradación, sino que sólo las secuestra en
el citoplasma evitando que puedan ingresar al núcleo para activar los genes
de respuesta al interferón (Rodríguez y col., 2002).
En el caso particular del RVP
no se ha investigado el papel que pueden jugar las proteínas V y C en los procesos
de inhibición de la respuesta antiviral mediada por interferón; sin embargo,
la identificación de un desequilibrio de la proporción de linfocitos CD4:CD8
y la proliferación predominante de linfocitos T CD4+CD8+ durante la infección,
estos últimos favorecedores de una respuesta inmune de tipo TH2, por su elevada
producción de IL10 y escaso IFNγ
(Hernández y col., 1998, 2001, 2002),
sugieren que alguna de las proteínas virales no estructurales está jugando un
papel en este proceso. Estos mecanismos son muy importantes en la patogenia
viral porque la inactivación de la respuesta a interferón podría permitir la
supervivencia del virus en el organismo, lo cual, aunado a otros procesos como
la persistencia y la falta de una vacuna efectiva, pueden estar involucrados
en los problemas de control de la enfermedad.
PERSPECTIVAS EN LA INVESTIGACION
SOBRE RVP
Es conocida la alta frecuencia
de mutación de los virus ARN, en la que se han propuesto como factores determinantes:
la relativa falta de fidelidad de las polimerasas virales, la presión selectiva
producida por el sistema inmunológico de los organismos hospederos, los cambios
en el medio ambiente, los eventos de recombinación y otros factores que han
sido descritos por múltiples autores (Holland y col., 1982;
Morse y Schluederberg, 1990; Nichol y col.,
1993; Murphy, 1994; Domingo y Holland,
1997; Aranda y col., 1997). Los virus ARN son quizá los
más variables entre todas las entidades autorreplicantes de la naturaleza, llegando
a registrar tasas de mutación entre 10-3 y 10-5 por genoma
por replicación (Domingo, 1989; Holland y
col., 1982; Drake, 1993). Existe una amplia bibliografía
respecto a la aparición de nuevas variantes y cepas de diversas especies de
la familia Paramyxoviridae (Iorio y col., 1986;
Smith e Inglis 1987; Hetherington y col.,
1994; Wild y Buckland, 1994; Kashiwagi
y col., 1997; Yates y col., 1997; Heinen
y col., 1998). Cada vez son más frecuentes los reportes que anuncian el
reconocimiento y caracterización de nuevas cepas y tipos virales, que pueden
llegar a ocasionar problemas de salud (Smith e Inglis 1987;
Domingo, 1989; De Jong y col., 1995),
y es precisamente en este aspecto en que se hace patente la necesidad de contar
con modelos para el estudio de la evolución de los virus, así como para entender
la evolución de los seres vivos en general, a nivel molecular (Becker,
1996; Domingo y Holland, 1997). Las herramientas moleculares
para el estudio de la evolución viral han aportado opciones para la modificación
de terapias antivirales y estrategias preventivas mediante vacunas constituidas
por múltiples componentes virales (Domingo, 1989; Novella
y col., 1995; Taboga y col., 1997).
Como se ha descrito en el texto,
las proteínas de los paramixovirus, y en particular del RVP, realizan distintas
funciones que impactan de diversas maneras el curso de la infección. El conocimiento
integral de la estructura y función de las proteínas virales permitirá diseñar
estrategias adecuadas para el control de la enfermedad.
AGRADECIMIENTOS
Parte de los datos reportados
en esta revisión fueron aportados por trabajos con financiamiento del Consejo
Nacional de Ciencia y Tecnología (Proyecto 3228PB9607). Los autores de esta
revisión fueron becarios de maestría y doctorado del Consejo Nacional de Ciencia
y Tecnología.
REFERENCIAS
ALLAN, G.M.,
F. MACNEILLY, Y. WALKER, T. LINNE, J. MORENO-LOPEZ, P. HERNANDEZ, S. KENNEDY,
B.P. CARROL, B. HERRON, J.C. FOSTER, B. ADAIR. 1996. A sequential study of experimental
porcine paramyxovirus (LPMV) infection of pigs: immunostaining of cryostat sections
and virus isolation. J. Vet. Diagn. Invest.
8: 405-413.
ARANDA, M.A.,
A. FRAILE, J. DOPAZO, J. M. MALPICA, F. GARCIA-ARENAL. 1997. Contribution of
mutation and RNA recombination to the evolution of a plant patogenic RNA. J.
Mol. Evol. 44: 81-88.
BECKER, Y. 1996.
A short introduction to the origin and molecular evolution of viruses. Virus
Genes. 11: 73-77.
BERG, M., A.
SUNDQVIST, J. MORENO-LOPEZ, T. LINNE. 1991. Identification of the porcine paramyxovirus
LPMV matrix protein gene: comparative sequence analysis with other paramyxoviruses.
J. Gen. Virol. 72: 1045-1050.
BERG, M., A.-C.
BERGVALL, M. SVENDA, A. SUNDQVIST, J. MORENO-LOPEZ, T. LINNE. 1997. Analysis
of the fusion protein gene of the porcine rubulavirus LPMV: comparative analysis
of paramyxoviruses F proteins. Virus Genes. 14: 55-61.
BERG, M., B.
HJERTNER, J. MORENO-LOPEZ, T. LINNE. 1992. The P gene of the porcine paramyxovirus
LPMV encodes three possible polipeptides, P, V and C, the P protein mRNA is
edited. J. Gen. Virol. 73: 1195-1200.
BERGER, E.A.,
P.M. MURPHY, J.M. FARBER. 1999. Chemokine receptors as HIV-1 coreceptors: roles
in viral entry tropism, and disease. Annu. Rev. Immunol.
17: 657-700.
CALAIN, P.,
L. ROUX. 1993. The rule of six, a basic feature for efficient replication of
Sendai virus defective interfering RNA. J. Virol. 67: 4822-4830.
CAMPOS, H.R.,
S.M. CARVAJAL. 1989. Trastornos reproductivos en los sementales de una granja
porcina de ciclo completo ante un brote de ojo azul. Memorias de la XXVI Reunión
Nacional de AMVEC, Manzanillo, Col., México, pp. 62-64.
CELMA, M.L.,
R. FERNANDEZ-MUÑOZ. 1992. Measles virus gene expression in lytic and persistent
infections of human lymphoblastoid cell line. J. Gen. Virol.
73: 2203-2209.
CUEVAS-ROMERO,
J.S. 2002. Persistence of porcine rubulavirus (PoRV-LPMV) in naturally infected
pigs. International Master of Sciences Programme, Faculty of Veterinary Medicine,
Swedish University of Agricultural Sciences, Uppsala.
CURRAN, J.,
R. BOECK, D. KOLAKOFSKY. 1991. The Sendai virus P gene expresses both an essential
protein and an inhibitor or RNA synthesis by shuffling modules via mRNA editing.
EMBO J. 10: 3079-3085.
CURRAN, J.,
J.B. MARQ, D. KOLAKOFSKY. 1992. The Sendai virus nonstructural C proteins specifically
inhibit viral mRNA synthesis. Virology 189: 647-656.
DE JONG, M.D.,
C.A.B. BOUCHER, G.J. GALASSO, M.S. HIRSH, E.R. KERN. 1995. Consensus symposium
on combined antiviral therapy. Antiviral Res. 29: 5-29.
DENG, R.,
Z. WANG, P.J. MAHON, M. MARINELLO, A.M. MIRZA, R.M. IORIO. 1995. Mutations in
the NDV HN protein that interfere with its ability to interact with the homologous
F protein in the promotion of fusion. Virology 253: 43-54.
DIDCOCK, L.,
D.F. YOUNG, S. GOODBOURN, R.E. RANDALL. 1999a. The V protein of simian virus
5 inhibits interferon signalling by targeting STAT1 for proteasome-mediated
degradation. J. Virol. 73(12): 9928-33.
DIDCOCK, L.,
D.F. YOUNG, S.GOODBOURN, R.E. RANDALL. 1999b. Sendai virus and simian virus
5 block activation of interferon-responsive genes: importance for virus pathogenesis.
J. Virol. 73: 3125-33.
DOMINGO, E.
1989. RNA virus evolution and the control of viral disease. Prog. Drug.
Res. 33: 93-133.
DOMINGO, E,
J.J. HOLLAND. 1997. RNA virus mutations and fitness for survival. Annu.
Rev. Microbiol. 51: 151-178.
DRAKE, J.W.
1993. Rates of spontaneous mutation among RNA viruses. Proc. Natl.
Acad. Sci. USA. 90: 4171-4175.
EBATA, S.N.,
M.J. COTE, C. KANG, K. DIMOCK. 1991. The fusion and hemagglutinin-neuraminidase
glycoproteins of human parainfluenza virus 3 are both required for fusion. Virology.
83: 437-441.
FUENTES, R.M.,
N.R. CARREON, M.H. RAMIREZ, M.E. TRUJILLO, B.I. FRAIRE. 1992. Estudio piloto
de la frecuencia de anticuerpos contra el paramixovirus del ojo azul en cerdos
de la República Mexicana. Vet. Mex. 23: 37.
GOODBOURN,
S., L. DIDCOCK, R.E. RANDALL. 2000. Interferons: cell signalling, immune modulation,
antiviral response and virus countermeasures. J. Gen. Virol.
81(Pt 10): 2341-64.
HAANES, E.J.,
P. GUIMOND, R. WARDLEY. 1997. The bovine parainfluenza virus type-3 (BPIV-3)
hemagglutinin/neuraminidase glycoprotein expressed in baculovirus protects calves
against experimental BPIV-3 challenge. Vaccine. 15: 730-738.
HEINEN, E.,
W. HERBST, N. SCHMEER. 1998. Isolation of a cytopathogenic virus from a case
of porcine reproductive and respiratory syndrome (PRRS) and its characterization
as a parainfluenza virus type 2. Arch. Virol. 143: 2233-2239.
HERCYK, N,
S.M. HORIKAMI, S.A. MOYER. 1988. The vesicular stomatitis virus L-protein possesses
the mRNA methyltransferase activities. Virology. 163: 222-225.
HERNANDEZ,
J., Y. GARFIAS, A. NIETO; C. MERCADO, L.F. MONTANO, E. ZENTENO. 2001. Comparative
evaluation of the CD4+CD8+ and CD4+CD8- lymphocytes in the immune response to
porcine rubulavirus. Vet. Immunol. Immunopathol.
79(3-4): 249-59.
HERNANDEZ,
J., Y. GARFIAS, J. REYES-LEYVA, R. CHAVEZ, R. LASCURAIN; J. VARGAS, E. ZENTENO.
2002. Peanut and Amaranthus leucocarpus lectins discriminate
between memory and naive/quiescent porcine lymphocytes. Vet. Immunol.
Immunopathol. 84 (1-2): 71-82.
HERNANDEZ,
J., J. REYES-LEYVA, R. ZENTENO, H. RAMIREZ, P. HERNANDEZ-JAUREGUI, E. ZENTENO.
1998. Immunity to porcine rubulavirus infection in adult swine. Vet.
Immunol. Immunopathol. 64: 367-381.
HERNANDEZ,
J., J. REYES-LEYVA, H. RAMIREZ, O. VALENZUELA, E. ZENTENO. 2004. Características
de la respuesta inmune de cerdos infectados con el rubulavirus porcino. Rev.
Vet. Méx. En prensa.
HERNANDEZ-JAUREGUI,
P. 2003. The pathogenesis of the porcine rubulavirus (LPMV): Infection in PK-15
cultured cells and in experimentally infected pigs. Doctoral thesis Swedish
University of Agricultural Sciences Uppsala. Acta Universitatis Agriculturae
Sueciae. Veterinaria 155.
HERNANDEZ-JAUREGUI,
P., A. SUNDQVIST, M. FUENTES, A. DIAZ, J. REYES-LEYVA, E. HERNANDEZ, J. MORENO-LOPEZ.
1992. Correlación entre las pruebas de virus neutralización, inhibición de la
hemoaglutinación y ELISA en sueros vacunales y de brote para anticuerpos contra
el paramixovirus del Síndrome del Ojo Azul en cerdos. Vet. Mex.
23: 217-222.
HERNANDEZ-LOPEZ,
J., H. RAMIREZ-MENDOZA, R. ZENTENO-CUEVAS, J. MONROYBACILIO, J. REYES-LEYVA,
E. ZENTENO. 1997. Neumonitis inducida por el Rubulavirus porcino.
Rev. Inst. Nal. Enf. Resp. Méx.
10: 250-255.
HETHERINGTON,
S.V., A.S. WATSON, R.A. SCROGGS, A. PORTNER. 1994. Human parainfluenza virus
type 1 combines cocirculation of strains and development of geographicaly restrictes
linages. J. Infect. Dis. 169: 248-252.
HJERTNER,
B. 1997. Molecular and biological aspects of porcine rubulavirus (LPMV). Lytic
and persistent infections. Doctoral thesis Swedish University of Agricultural
Sciences Uppsala. Acta Universitatis Agriculturae Sueciae. Veterinaria
HJERTNER, B,
T. LINNE, J. MORENO-LOPEZ. 1998a. Establishment and characterisation of a porcine
rubulavirus (LPMV) persistent infection in porcine kidney cells. Acta.
Vet. Scand. 38: 213-224.
HJERTNER, B,
A.C. WIMAN, M. SVENDA, M. BERG, J. MORENO-LOPEZ, T. LINNE. 1998b. Multiple factors
including subgenomic RNAs and reduced viral protein expression are associated
with a persistent infection by porcine rubulavirus (LPMV). Arch. Virol.
143: 425-439.
HOLLAND, J,
K. SPINDLER, F. HORODYSKI, E. GRABAU, S. NICHOL, S. VANDEPOL. 1982. Rapid evolution
of RNA genomes. Science 215: 1577-1585.
HU, X, R. RAY,
R.W. COMPANS. 1992. Functional interactions between the fusion protein and hemagglutinin-
neuraminidase of human parainfluenza viruses. J. Virol. 66:
1528-1534.
HUMMEL, K.B.,
J.A. VANCHIERE, W.J. BELLINI. 1994. Restriction of fusion protein mRNA as a
mechanism of measles virus persistence. Virology 202: 665-672.
HUNT, D.M.,
K.L. HUTCHINSON. 1993. Amino acid changes in the L polymerase protein of vesicular
stomatitis virus which confer aberrant polyadenylation and temperature-sensitive
phenotypes. Virology 193: 786-793.
IORIO, R.M.,
J.B. BORGMAN, R.L. GLICKMAN, M.A. BRATT. 1986. Genetic variation within a neutralizing
domain on the haemagglutininneuraminidase glycoprotein of Newcastle disease
virus. J. Gen. Virol. 67: 1393-1403.
KASHIWAGI,
Y., H. KAWASHIMA, K. TAKEKUMA, A. HOSHIKA, T. MORI, T. NAKAYAMA. 1997. Detection
of mumps virus genome directly from clinical samples and a simple method for
genetic differentiation of the Hoshino vaccine strain from wild strains of mumps
virus. J. Med. Virol. 52: 195-199.
KATO, A., K.
KIYOTANI, Y. SAKAI, T. YOSHIDA, Y. NAGAI. 1997. The paramyxovirus, Sendai virus,
V protein encodes a luxury function required for viral pathogenesis. EMBO
J. 16: 578-587.
KATZ, M. 1994.
Clinical spectrum of measles. Curr. Top. Microbiol.
Immunol. 191: 1-12.
KOLAKOFSKY,
D., T. PELET, D. GARCIN, S, HAUSMANN, J. CURRAN, L. ROUX. 1998. Paramyxovirus
RNA synthesis and requeriment for hexamer genome length: the rule of six revisited.
J. Virol. 72: 891-899.
KUBOTA, T.,
N. YOKOSAWA, S. YOKOTA, N. FUJII. 2001. C terminal CYS-RICH region of mumps
virus structural V protein correlates with block of interferon alpha and gamma
signal transduction pathway through decrease of STAT 1-alpha. Biochem.
Biophys. Res. Commun. 283(1): 255-9.
LAMB, R.A.
1993. Paramyxovirus fusion: a hipothesis for changes. Virology 197:
1-11. LAMB, R.A., D. KOLAKOFSKY. 1996. Paramyxoviridae: The viruses and their
replication. In: B.N. Fields, D.M. Knipe, P.M. Howley y col. (ed.). Fields Virology.
Third ed. Lippincott- Raven Publishers, E.U.A.
LAMB, R.A.,
D. KOLAKOFSKY. 1996. Paramyxoviridae: The viruses and their replication. In:
B.N. Fields, D.M. Knipe, P.M. Howley y col. (ed.). Fields Virology. Third ed.
Lippincott- Raven Publishers, E.U.A.
LAZZARINI,
R.A., J.D. KEENE, M. SCHUBERT. 1981.The origins of defective interfering particles
of the negative-strand RNA viruses. Cell. 26: 145-154.
LINNE, T.,
M. BERG, A.-C. BERGVALL, B. HJERTNER, J. MORENO-LOPEZ. 1992. The molecular biology
of the porcine paramyxovirus LPMV. Vet. Microbiol. 33: 263-273.
MARKWELL, M.A.,
C.F. FOX. 1980. Protein-protein interactions within paramyxoviruses identified
by native disulfide bonding or reversible chemical cross-linking. J.
Virol. 33: 152-166.
MAYO MA. 2002.
A summary of taxonomic changes recently approved by ICTV. Arch. Virol.
147(8): 1655-63.
MCNEILLY,
F., Y. WALKER, G.M. ALLAN, J.C. FOSTER, T. LINNE, M. MERZA, P. HERNANDEZ, S.
KENNEDY, B. ADAIR. 1997. A comparative study on the use of virus and antibody
detection techniques for the diagnosis of La Piedad Michoacan paramyxovirus
(LPMV) infection in Pigs. J. Vet. Diagn. Invest.
9: 3-9.
MORENO-LOPEZ,
J., P. CORREA-GIRON, A. MARTINEZ, A. ERICSSON. 1986. Characterization of a paramyxovirus
isolated from the brain of a piglet in Mexico. Arch. Virol.
91: 221-231.
MORRISON,
T.G., A. PORTNER. 1991. Structure, function and intracellular processing of
glycoproteins of Paramyxoviridae, pp. 347-382. En: D.W. Kingsbury (ed.)
The Paramyxoviruses. Plenum Press, E.U.A.
MORSE, S.S.,
A. SCHLUEDERBERG. 1990. Emerging viruses: the evolution of viruses and viral
diseases. J. Infect. Dis. 162: 1-7.
MOSCONA, A.,
R.W. PELUSSO. 1991. Fusion properties of cells persistently infected with human
parainfluenza virus type 3: participation of hemagglutinin- neuraminidase in
membrane fusion. J. Virol. 65: 2773-2777.
MURPHY, D.G.,
K. DIMOCK, C.Y. KANG. 1987. Defective interfering particles of human parainfluenza
virus 3. Virology 158: 439-443.
MURPHY, F.A.
1994. New, emerging, and reemerging infectious diseases. Adv. Virus
Res. 43: 1-52.
NICHOL, S.T.,
C.F. SPIROPOULOU, S. MORZUNOV, P.E. ROLLIN, T.G. KSIAZEK, H. FELDMAN, A. SANCHEZ,
J. CHILDS, S. ZAKI, C.J. PETERS. 1993. Genetic identification of a Hantavirus
associated with an outbreak of acute respiratory illness. Science 262:
914-917.
NISHIO, M.,
D. GARCIN, V. SIMONET, D. KOLAKOFSKY. 2002. The carboxyl segment of the mumps
virus V protein associates with Stat proteins in vitro via a tryptophan-rich
motif. Virology 300(1): 92-9.
NOVELLA, I.S.,
E. DOMINGO, J.J. HOLLAND. 1995. Rapid viral quasispecies evolution: implications
for vaccine and drug strategies. Mol. Med. Today
1: 248-253.
RAMIREZ-HERRERA,
M.A., M.L. MENDOZ-AMAGAÑA, S.H. DUEÑAS. 1997. Experimental infection of swine
and cat central nervous systems by the pig paramyxovirus of the blue eye disease.
J. Vet. Med. B44: 461-476.
RAMIREZ-MENDOZA,
H., P. HERNANDEZ-JAUREGUI, J. REYES-LEYVA, E. ZENTENO, J. MORENO-LOPEZ, S. KENNEDY.
1997. Lesions in the reproductive tract of boars experimentally infected with
porcine rubulavirus. J. Comp. Path. 117: 237-252.
RAY, R., R.W.
COMPANS. 1990. Paramyxoviruses. In: M.H.B. Van Regenmortel, A.R. Neurath (ed.).
Immunochemistry of viruses II. The basis for serodiagnosis and vaccines. Elsevier,
New York.
REYES-LEYVA,
J, B. ESPINOSA, J. HERNANDEZ, R. ZENTENO, V. VALLEJO, P. HERNANDEZJAUREGUI,
E. ZENTENO. 1997. Neua2,3-Gal glycoconjugate expresion determines cell susceptibility
to the porcine rubulavirus LPMV. Comp. Biochem. Physiol.
118B: 327-332.
REYES-LEYVA,
J, P. HERNANDEZ-JAUREGUI, L.F. MONTAÑO, E. ZENTENO. 1993. The porcine paramyxovirus
LPM specifically recognizes sialyl(a2,3) lactose-containing structures. Arch.
Virol. 133: 195-200.
REYES-LEYVA,
J., B. ESPINOSA, G. SANTOS, R. ZENTENO, J. HERNANDEZ, V.VALLEJO, E. ZENTENO.
1999. Purification and characterization of the Hemagglutinin-neuraminidase of
Porcine Rubulavirus LPMV. Glycoconj. J.
16: 517-522.
REYES-LEYVA,
J., O. GARCIA MORALES, G. SANTOS-LOPEZ, V. VALLEJO-RUIZ, H. RAMIREZ- MENDOZA,
J. HERNANDEZ. 2004. Detección de viremia en la infección por Rubulavirus porcino.
Arch. Med. Vet. En proceso de revisión.
REYES-LEYVA,
J., G. SANTOS, J. HERNANDEZ, B. ESPINOSA, M.T. BORRAZ, H. RAMIREZ, V. VALLEJO,
E. ZENTENO. 2002. Mecanismos moleculares de la patogenia viral: Estudios con
el Rubulavirus porcino. En: Cea Bonilla A., del Arenal Mena
IP, Riveros Rosas H, Vázquez Contreras E (eds). Mensaje Bioquímico, Vol. XXVI.
Depto. Bioquímica, Fac. Medicina, Universidad Nacional Autónoma de México. México,
D.F., México.
RIMA, B., D.J.
ALEXANDER, M.A. BILLETER, P.L. COLLINS, D.W. KINGSBURY, M.A. LIPKIND, Y. NAGAI,
C, ÖRVELL, C.R. PRINGLE, V. TER MULLEN. 1995. Family Paramyxoviridae, p. 265-274.
In: F.A. Murphy, C.M. Fauquet, D.H.L. Bishop, S.A. Ghabrial, A.W. Jarvis, G.P.
Matelli, M.A. Mayo, M.D. Summers (ed.). Virus taxonomy. Classification and nomenclature
of viruses. Viena-Nueva York: Springer-Verlag.
RIMA, B.K.,
J.A.P. EARLE: K. BACZKO, P.A. ROTA, W.J. BELLINI. 1994. Measles virus strain
variation. Curr. Top. Microbiol. Immunol.
191: 65-83.
RODRIGUEZ,
J.J., J.P. PARISIEN, C.M. HORVATH. 2002. Nipah Virus V Protein Evades Alpha
and Gamma Interferons by Preventing STAT1 and STAT2 Activation and Nuclear Accumulation.
J. Virol. 76(22): 11476-83.
ROSALES, E.F.,
L.A. MARTINEZ, G.P. CORREA. 1988. Análisis retrospectivo (1972-1986) de la presencia
de anticuerpos inhibidores de la hemaglutinación contra el paramixovirus porcino
LPM, al parecer asociado con el "Síndrome de Ojo Azul". Tec. Pec.
26: 304-311.
SANCHEZ, A.,
B.P. DE, A.K. BANERJEE. 1985. In vitro phosphorylation of NS protein by the
L protein of vesicular stomatitis virus. J. Gen. Virol. 66:
1025-1036.
SANDERSON,
C.M., H.H. WU, D.P. NAYAK. 1993. Sendai virus M protein binds independently
to either the F or the HN glycoprotein in vivo. J. Virol. 68: 69-76.
SANTOS-LOPEZ,
G., E. FLORES, R. BAÑOS, I. HERRERA-CAMACHO, J. REYES-LEYVA. 2004. Purification
of the porcine rubulavirus attachment protein by liquid isoelectric focusing.
Protein Expr. Purif. En prensa.
SERGEL, T.,
L.W. MACGUINNES, M.E. PEEPLES, T.G. MORRISON. 1993. The attachment function
of the Newcastle disease virus hemagglutininneuraminidase protein can be separated
from fusion promotion by mutation. Virology 193: 717-726.
SHEID, A.,
P.W. CHOPPIN. 1974. Identification of biological activities of paramyxovirus
glycoproteins. Activation of cell fusion, hemolysis, and infectivity of proteolytic
cleavage of an inactive precursor protein of Sendai virus. Virology
57: 475-490.
SMITH, D. B.,
S.C. INGLIS. 1987. The mutation rate and variability of eucaryotic viruses:
an analytical review. J. Gen. Virol. 68: 2729-2740.
SMITH, G. L.
1994. Virus strategies for evasion of the host response to infection. Trends
Microbiol. 2: 81-88.
STARK, G.R.,
I.M. KERR, B.R. WILLIAMS, R.H. SILVERMAN, R.D. SCHREIBER. 1998. How cells respond
to interferons. Annu. Rev. Biochem. 67: 227-64.
STEPHANO, H.A.
1999. Blue eye disease. In: B.E. Straw, S. D'Allaire, W.L. Mengelin, D.J. Taylor
(eds.). Diseases of swine. 8th ed. Ames, Iowa. Iowa State University Press.
Pp 103-112.
STEPHAN, H.A.,
G.M. GAY, T.C. RAMIREZ. 1988. Encephalomyelitis, reproductive failure and corneal
opacity (blue eye) in pigs, associate with a paramyxovirus infection. Vet.
Rec. 122: 6-10.
STONE-HULSLANDER,
J., T.G. MORRISON. 1999. Mutational analysis of heptad repeats in the membrane-
proximal region of Newcastle disease virus HN protein. J. Virol.
73: 3630-3637.
SUNDQVIST,
A., M. BERG, P. HERNANDEZJAUREGUI, T. LINNE, J. MORENO-LOPEZ. 1990. The structural
proteins of a porcine paramyxovirus (LPMV). J. Gen. Virol.
71: 609-613.
SUNDQVIST,
A., M. BERG, P. HERNANDEZJAUREGUI, T. LINNE, J. MORENO-LOPEZ. 1991. The haemaglutinin-neuraminidase
gene of the porcine paramyxovirus LPMV: comparison with other paramyxovirus
revealed the closest to simian virus 5 and mumps virus. Arch. Virol.
122: 331-340.
SVENDA, M.,
M. BERG, J. MORENO-LOPEZ, T. LINNE. 1997. Analysis of the large (L) protein
gene of the porcine rubulavirus LPMV: identification of possible functional
domains. Virus Res. 48: 57-70.
SVENDA, M.,
B. HJERTNER, T. LINNE, M. BERG. 2002. Both the P and V proteins of the porcine
rubulavirus LPMV interact with the NP protein via their respective C-terminal
unique parts. Virus Res. 83(1-2): 31-41.
TABOGA, O.,
C. TAMI, E. CARRILLO, J.I. NUÑEZ, A. RODRIGUEZ, J.C. SAIZ, E. BLANCO, M.L. VALERO,
X. ROIG, J.A. CAMARERO, D. ANDREU, M.G. MATEU , E. GIRALT, E. DOMINGO, F. SOBRINO,
E. PALMA. 1997. A large-scale evaluation of peptide vaccines against foot-and-mouth
disease: lack of solid protection in cattle and isolation of scape mutants.
J. Virol. 71: 2606-2614.
TSURUDOME,
M., M. KAWANO, T. YUASA, M. NISHIO, K. HIROSHI, Y. ITO. 1995. Identification
of regions on the hemagglutininneuraminidase protein of human parainfluenza
virus type 2 important for promoting cell fusion. Virology 213: 190-203.
VALLEJO V.,
J. REYES-LEYVA, J. HERNANDEZ, H. RAMIREZ, P. DELANNOY, E. ZENTENO. 2000. Differential
expression of sialic acid on porcine organs during maturation process. Comp.
Biochem. Physiol. Part B 126: 415-424.
WHITE, J.M.
1990. Viral and cellular membrane fusion proteins. Annu. Rev.
Physiol. 52: 675-697.
WILD, T.F.,
R. BUCKLAND. 1994. Functional aspects of envelope-associated measles virus proteins.
Curr. Top. Microbiol. Immunol. 191: 51-64.
WIMAN, AC,
B. HJERTNER, T. LINNE, G. ALLAN, F. MCNEILLY, J. MORENO-LOPEZ, M. BERG. 1998.
Porcine rubulavirus LPMV RNA persists in the central nervous system of pigs
after recovery from acute infection. J. Neurovirol. 4: 545-552.
WOLINSKY, J.S.
1996. Mumps virus. In: B.N. Fields, D.M. Knipe, P.M. Howley y col. (ed.) Fields
Virology. Trd ed. Lippincott-Raven Publishers, E.U.A.
YATES, P.J.,
M.A. AFZAL, P.D. MINOR. 1997. Antigenic and genetic variation of the HN protein
of mumps virus strains. J. Gen. Virol. 77: 2491-2497.
ZENTENO-CUEVAS,
R., J. HERNANDEZ, B. ESPINOSA, J. REYES-LEYVA, E. ZENTENO. 1998. Secondary structure
prediction of the hemagglutinin-neuraminidase from a porcine rubulavirus. Arch.
Virol. 143: 333-352.
Aceptado: 23-03-2004.


